Comunicación corta
Biotecnología Vegetal Vol. 18, No. 1: 57 - 60, enero - marzo, 2018
Instituto de Biotecnología de las Plantas. UCLV. MES.
eISSN 2074-8647, RNPS: 2154
Perfil de expresión de una lipasa de tipo GDSL durante interacciones compatible e incompatible entre Musa spp. y Pseudocercospora fijiensis
Expression profile of a GDSL-like lipase during compatible and incompatible interactions between Musa spp. and Pseudocercospora fijiensis
Mairenys Concepción-Hernández1, Mayra Acosta-Suárez1, Milady Mendoza-Rodríguez1, Orelvis Portal2
1Instituto de Biotecnología de las Plantas, Universidad Central Marta Abreu de Las Villas. Carretera a Camajuaní km 5,5. Santa Clara. Villa Clara. Cuba. CP 54 830. e-mail: mairenys@ibp.co.cu
2Departamento de Biología, Facultad de Ciencias Agropecuarias, Universidad Central Marta Abreu de Las Villas. Carretera a Camajuaní km 5,5. Santa Clara. Villa Clara. Cuba. CP 54 830.
RESUMEN
La identificación de genes de interés para el mejoramiento genético de Musa spp. contra Pseudocercospora fijiensis Morelet se ve limitada por el escaso conocimiento de los mecanismos moleculares de la interacción planta-patógeno. Las lipasas de tipo GDSL constituyen blancos promisorios al estar relacionadas con los mecanismos de resistencia y suceptibilidad a patógenos. El objetivo de este trabajo fue determinar mediante PCR cuantitativo los perfiles de expresión del gen de una lipasa de tipo GDSL de Musa acuminata Colla durante las interacciones compatible e incompatible con P. fijiensis. A partir del análisis realizado se identificó el gen que codifica para la lipasa de tipo GDSL en el genoma de banano y se comprobó su sobreexpresión en la interacción compatible en estadio temprano de infección.
Palabras clave: Mycosphaerella fijiensis, PCR cuantitativo, Sigatoka negra
ABSTRACT
The identification of genes of interest for the genetic improvement of Musa spp. against Pseudocercospora fijiensis Morelet is constrained by the limited knowledge of the molecular mechanisms of the plant-pathogen interaction. GDSL-like lipases are promising targets since they are related to the mechanisms of resistance and susceptibility to pathogens. The aim of this work was to determine, through quantitative PCR, the expression profiles of a GDSL-like lipase gene from Musa acuminata Colla during compatible and incompatible interactions with P. fijiensis. Based on this analysis, the gene coding for the GDSL-like lipase was identified in the banana genome and its overexpression was verified in the compatible interaction at the early stage of infection.
Keywords: black Sigatoka, Mycosphaerella fijiensis, quantitative PCR
INTRODUCCIÓN
El cultivo de bananos y plátanos (Musa spp.) a nivel mundial se ve afectado por la incidencia de enfermedades y plagas, dentro de las que destaca la Sigatoka negra, causada por Pseudocercospora fijiensis (Morelet) Deighton. La transformación genética constituye una alternativa para obtener cultivares resistentes a esta enfermedad. Sin embargo, no se cuenta con cultivares de bananos comerciales obtenidos por esta técnica con resistencia a P. fijiensis, por lo que la búsqueda de nuevos genes y estrategias continúa.
Portal et al. (2011) identificaron un grupo de EST (del inglés: Expressed Sequence Tag) que se expresaban de forma diferencial durante la interacción compatible ‘Grande naine’ (Musa AAA)- P. fijiensis. Uno de estos genes fue identificado como una posible lipasa de tipo GDSL mediante BLASTX por similitud de secuencias con la proteína AAP05801.1 de arroz (Oryza sativa L.).
En otros patosistemas, las lipasas de tipo GDSL se han relacionado con la actividad antifúngica. Por ejemplo, la lipasa de tipo GDSL de Arabidopsis thaliana (L.) Heynh. AtGLIP1 modula la resistencia a Alternaria brassicicola (Schwein.) Wiltshire al provocar la ruptura de las hifas y esporas del hongo (Oh et al., 2005), y a través de la inducción de una respuesta de defensa mediada por etileno (Kwon et al., 2009). Además, en pimiento (Capsicum annum L.) CaGLIP1 está involucrada en la respuesta de defensa al daño y a patógenos (Hong et al., 2008). Adicionalmente, AtGLIP2 participa en la defensa contra Erwinia carotovora (Jones) Dye mediante la regulación negativa de la vía de señalización por auxinas (Lee et al., 2009).
El objetivo de este trabajo fue determinar mediante PCR cuantitativo los perfiles de expresión del gen de una lipasa de tipo GDSL de Musa acuminata Colla durante las interacciones compatible e incompatible. A partir del análisis realizado se identificó el gen que codifica para dicha lipasa en el genoma de banano y se comprobó su sobreexpresión en la interacción compatible en estadio temprano.
MATERIALES Y MÉTODOS
Las dos secuencias clasificadas previamente como lipasas de tipo GDSL, a partir de la biblioteca sustractiva de ADNc (Portal et al., 2011) fueron comparadas mediante BLAST con la base de datos del genoma de M. accuminata (D’hont et al., 2012), a través de la plataforma Banana Genome Hub (http://banana-genome-hub.southgreen.fr/), con el objetivo de identificar el gen correspondiente en Musa spp.
Las plantas de 'Calcutta 4' y 'Grande naine' se propagaron in vitro y se cultivaron durante tres meses en condiciones de casa de cultivo (Orellana, 1994). La inoculación con una suspensión micelial de P. fijiensis (cepa CCIBP-Pf-83) se realizó de acuerdo con el protocolo descrito por Leiva-Mora et al. (2010).
El ARN total se aisló a partir de hojas infectadas y no infectadas de acuerdo con el protocolo descrito por Liao et al. (2004). Se tomaron tres réplicas biológicas en cada tiempo de muestreo (3, 6, 9, 12 y 15 días posteriores a la inoculación; dpi).
La amplificación por PCR cuantitativo se realizó según lo descrito por Portal et al. (2011) en placas ópticas de 96 pocillos con el sistema de detección en tiempo real Mx3005P (Stratagene) con la mezcla maestra SYBR Green (Stratagene). Para la amplificación de la lipasa de tipo GDSL se emplearon los cebadores siguientes: Gdsl_Fwd: 5’-CCCATCACAAAAGAAGAAG-3’ y Gdsl_Rev: 5’-CCCCGTCAACATTCATCAG-3’ y para el gen constitutivo de referencia actina I de M. acuminata (GenBank EF672732.1) Act1_Fwd: 5’-GGTTAGACATCCTTTTCCTCTC-3’ y Act1_Rev: 5’- GCATCATCTCCAGCGAAAC-3’, que generan amplicones de 120 pb y 130 pb, respectivamente. Los niveles de expresión del gen se expresaron como relativos a los niveles en plantas no infectadas (Control) en cada tiempo de muestreo.
RESULTADOS Y DISCUSIÓN
La comparación de las secuencias de Portal et al. (2011) con la base de datos del genoma de Musa accuminata mostró que los dos EST obtenidos en la biblioteca sustractiva correspondieron a un único gen, identificado como Ma02_g12980 y anotado como lipasa de tipo GDSL (D’hont et al., 2012).
Este gen está localizado en el cromosoma 2, y consta de cinco exones distribuidos en 1591 pb y codifica para un polipéptido de 356 aa, según la información disponible en Banana Genome Hub (Droc et al., 2013).
El análisis realizado mostró una sobreexpresión del gen de la lipasa de tipo GDSL en el cultivar susceptible a los 3 dpi con P. fijiensis (Figura 1).
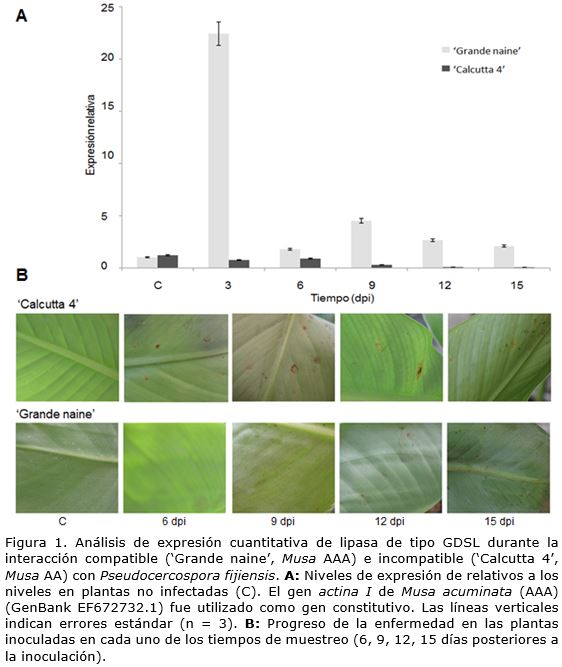
Por otro lado, en el cultivar resistente se reprimió la expresión del gen de la lipasa durante el período analizado (Figura 1). Este resultado podría indicar un posible papel de esta enzima en los procesos de susceptibilidad, en lugar de resistencia, al estar relacionada con una respuesta inadecuada ante la presencia del patógeno.
Al respecto, se ha descrito que la lipasa 1 de tipo GDSL (CaGLIP1) aumenta la susceptibilidad a Xanthomonas campestris pv. vesicatoria (Doidge) Dye. en pimiento, a Pseudomonas syringae pv. tomato van Hall 1902 y al oomicete biotrófico Hyaloperonospora parasitica (Pers.) Constant., cuando es sobreexpresada en A. thaliana (Hong et al., 2008). Adicionalmente en arroz, la represión simultánea de los genes de las lipasas de tipo GDSL, OsGLIP1 y OsGLIP2 aumenta la resistencia a patógenos fúngicos y bacterianos, mientras que su sobreexpresión incrementa la susceptibilidad (Gao et al., 2017). Tanto CaGLIP1, como OsGLIP1 y OsGLIP2 han sido identificados como reguladores negativos de la inmunidad, lo que sugiere una variabilidad de funciones dentro de este grupo de enzimas. Los estudios mencionados contribuyen a la acumulación de evidencias sobre el papel de los lípidos y sus productos en la inmunidad en plantas. Por tanto, el estudio de las lipasas de tipo GDSL como enzimas reguladoras del metabolismo lipídico podría resultar promisorio para el mejoramiento genético en plantas.
CONCLUSIONES
El gen Ma02_g12980 presente en el genoma de M. acuminata codifica para una lipasa de tipo GDSL y esta se expresa de manera diferencial durante las interacciones compatible e incompatible con P. fijiensis.
ACKNOWLEDGMENTS
Los autores agradecen el apoyo del VLIR-UOS a través de una estancia corta de investigación de Orelvis Portal en el Laboratorio de Fitopatología de la Facultad de Bioingeniería en la Universidad de Gante, Bélgica.
Conflictos de intereses
Los autores declaran que no existen conflictos de intereses.
REFERENCIAS
D’hont A, Denoeud F, Aury JM, Baurens FC, Carreel F, Garsmeur O, Da Silva C (2012) The banana (Musa acuminata) genome and the evolution of monocotyledonous plants. Nature 488(7410): 213; doi:10.1038/nature11241
Droc G, Lariviere D, Guignon V, Yahiaoui N, This D, Garsmeur O, Lengelle J (2013) The banana genome hub. Database 2013; doi:10.1093/database/bat035
Gao M, Yin X, Yang W, Lam SM, Tong X, Liu J, Wang X, Li Q, Shui G, He Z (2017) GDSL lipases modulate immunity through lipid homeostasis in rice. PLoS Pathogens 13(11): e1006724; doi:10.1371/journal.ppat.1006724
Hong JK, Choi HW, Hwang IS, Kim DS, Kim NH, Choi DS, Kim YJ, Hwang BK (2008) Function of a novel GDSL-type pepper lipase gene, CaGLIP1, in disease susceptibility and abiotic stress tolerance. Planta 227(3): 539-558; doi:10.1007/s00425-007-0637-5
Kwon SJ, Jin HC, Lee S, Nam MH, Chung JH, Kwon SI, Ryu CM, Park OK (2009) GDSL lipase‐like 1 regulates systemic resistance associated with ethylene signaling in Arabidopsis. The Plant Journal 58(2): 235-245; doi:10.1111/j.1365-313X.2008.03772.x
Lee DS, Kim BK, Kwon SJ, Jin HC, Park OK (2009) Arabidopsis GDSL lipase 2 plays a role in pathogen defense via negative regulation of auxin signaling. Biochemical and Biophysical Research Communications 379(4): 1038-1042; doi:10.1016/j.bbrc.2009.01.006
Leiva-Mora M, Alvarado-Capó Y, Acosta-Suárez M, Cruz-Martín M, Sánchez-García C, Roque B (2010) Protocolo para la inoculación artificial de plantas de Musa spp. con
Mycosphaerella fijiensis y evaluación de su respuesta mediante variables epifitiológicas y componentes de la resistencia. Biotecnología Vegetal 10(2): 79-88.
Liao Z, Chen M, Guo L, Gong Y, Tang F, Sun X, Tang K (2004) Rapid isolation of high quality total RNA from Taxus and Ginkgo. Preparative Biochemistry and Biotechnology 34(3): 209-214; doi:10.1081/PB-200026790
Oh IS, Park AR, Bae MS, Kwon SJ, Kim YS, Lee JE, Kang NY, Lee S, Cheong H, Park OK (2005) Secretome analysis reveals an Arabidopsis lipase involved in defense against Alternaria brassicicola. The Plant Cell 17(10): 2832-2847; doi:10.1105/tpc.105.034819
Orellana PP (1994) Tecnología para la micropropagación in vitro de clones de Musa spp. Tesis en opción del Grado Científico de Doctor en Ciencias Agrícolas, Universidad Central Marta Abreu de Las Villas, Santa Clara, Cuba
Portal O, Izquierdo Y, De Vleesschauwer D, Sánchez-Rodríguez A, Mendoza-Rodríguez M, Acosta-Suárez M, Ocaña B, Jiménez E, Höfte M (2011) Analysis of expressed sequence tags derived from a compatible Mycosphaerella fijiensis–banana interaction. Plant Cell Reports 30(5): 913-928; doi:10.1007/s00299-011-1008-z
Recibido: 17-10-2017
Aceptado: 06-12-2017
Copyright (c) 2018 Biotecnología Vegetal
Biotecnología Vegetal eISSN 2074-8647, RNPS: 2154. ISSN 1609-1841, RNPS: 0397 Editada por: Instituto de Biotecnología de las Plantas. Universidad Central Marta Abreu de Las Villas. Carretera a Camajuaní km 5.5, Santa Clara, Villa Clara, Cuba CP 54 830 Tel: 53 42200124, e-mail: info@ibp.co.cu

Biotecnología Vegetal está bajo una Licencia Creative Commons Atribución-NoComercial 4.0 Internacional.
