NOTA TÉCNICA
Biotecnología Vegetal Vol. 14, No. 3: 151 - 154, julio - septiembre,
2014
ISSN 2074-8647, RNPS: 2154 (Versión electrónica)
Instituto de Biotecnología de las Plantas. UCLV. MES.
Extracción in situ de ADN genómico para el análisis por PCR de regiones de interés en cuatro especies vegetales y un hongo filamentoso
In situ genomic DNA extraction for PCR analysis of regions of interest in four plant species and one filamentous fungi
Luis E. Rojas1*, Maritza Reyes1, Naivy Pérez-Alonso1, María I. Olóriz1, Laisyn Posada-Pérez1, Bárbara Ocaña1, Orelvis Portal2, Borys Chong-Pérez1, Jorge L. Pérez Pérez *Autor para correspondencia
1Instituto
de Biotecnología de las Plantas, Universidad Central Marta Abreu de Las
Villas. Carretera a Camajuaní km 5.5. Santa Clara. Villa Clara, Cuba.
CP 54 830. e-mail: luis@ibp.co.cu
2Departamento de Biología. Facultad de Ciencias Agropecuaria, Universidad Central Marta Abreu de Las Villas. Carretera a Camajuaní km 5.5. Santa Clara. Villa Clara, Cuba. CP 54 830.
RESUMEN
Los métodos de extracción de ADN genómico son generalmente trabajosos y peligrosos para la salud humana y el medio ambiente por el uso de solventes orgánicos (cloroformo y fenol). En este trabajo se valida un protocolo de extracción in situ de ADN genómico por lisis alcalina. Se empleó con el objetivo de amplificar regiones del ADN en cuatro especies de plantas y de un hongo mediante la reacción en cadena de la polimerasa (PCR). Se utilizó material vegetal de Saccharum officinarum L., Carica papaya L., Digitalis purpurea L. de los cuales se logró extender diferentes regiones del genoma a través de PCR. Fue posible amplificar un fragmento del gen avr-4 de ADN purificado a partir de micelio liofilizado de Mycosphaerella fijiensis Morelet. Adicionalmente, se logró amplificar la región del transgen ap24 insertado en el genoma de banano cv. `Grande naine' (Musa AAA).
Palabras clave: Carica papaya L., Digitalis purpurea L., lisis alcalina, Musa, Saccharum officinarum L.
ABSTRACT
The extraction methods of genomic DNA are usually laborious and hazardous to human health and the environment by the use of organic solvents (chloroform and phenol). In this work a protocol for in situ extraction of genomic DNA by alkaline lysis is validated. It was used in order to amplify regions of DNA in four species of plants and fungi by polymerase chain reaction (PCR). From plant material of Saccharum officinarum L., Carica papaya L. and Digitalis purpurea L. it was possible to extend different regions of the genome through PCR. Furthermore, it was possible to amplify a fragment of avr-4 gene DNA purified from lyophilized mycelium of Mycosphaerella fijiensis. Additionally, it was possible to amplify the region ap24 transgene inserted into the genome of banana cv. `Grande naine' (Musa AAA).
Key words: alkaline lysis, Carica papaya L., Digitalis purpurea L., Musa, Saccharum officinarum L.
INTRODUCCIÓN
Los métodos
de extracción de ácido desoxirribonucleico (ADN) han evolucionado
desde protocolos manuales extremadamente largos y fatigosos hasta los llamados
kit de extracción que son cómodos y rápidos para el que
los ejecuta. Estos sistemas de extracción tienen la desventaja de ser
costosos y por ello cuando se necesita hacer estudios moleculares a elevadas
cantidades de muestras, los costos pueden llegar a ser prohibitivos. Por estos
motivos muchos investigadores incursionan en protocolos más sencillos
y de fácil ejecución como son
los llamados sistemas de extracción in situ. Estos tienen varias
ventajas tales como: son rápidos y efectivos cuando se quiere extraer
ADN de pequeñas muestras frescas o liofilizadas, no se necesita el uso
de nitrógeno líquido ni el empleo de solventes orgánicos,
en poco tiempo se pueden procesar gran número de muestras y el costo
es mínimo (Xin et al., 2003).
El protocolo utilizado en este trabajo fue referido por Xin et al. (2003). Estos autores lo aplicaron en tabaco (Nicotiana tabacum L.), sorgo (Sorghum bicolor (L.) Moench), algodón (Gossypium arboreum L.), musgo (Polytrichum commune), Arabidopsis y pino (Pinus L.) con el objetivo de procesar grandes cantidades de muestras y se obtuvieron resultados satisfactorios en los PCR ejecutados posteriormente. Consta de un paso inicial de lisis alcalina con hidróxido de sodio y el detergente Tween 20. Posteriormente el pH de la solución se neutraliza con un tampón de Tris-HCl pH 2 y EDTA y así se logra el pH requerido para las reacciones de PCR. Este protocolo ha sido empleado por otros autores en diferentes especies vegetales (Pan, 2010; Hao et al., 2011). Sin embargo, no se ha informado de su uso para la extracción de ADN de Digitalis purpurea L., ni en hongos.
Este trabajo persiguió como objetivo validar el protocolo de extracción de ADN in situ en cuatro especies vegetales y un hongo filamentoso.
MATERIALES Y MÉTODOS
Material vegetal
Se emplearon hojas de plantas de banano cv. `Grande naine' (Musa AAA) transformado con el gen ap24 de tabaco que crecieron en casa de cultivo por tres meses, hojas de plantas de caña de azúcar (Saccharum officinarun L.) cv. `B-4362' cultivadas durante cinco meses en vivero, plantas in vitro de papaya (Carica papaya L.) cv. `Maradol rojo' en fase de multiplicación y plantas in vitro de Digitalis purpurea L. en fase de multiplicación.
Todas las plantas utilizadas procedían de material vegetal propagado por cultivo in vitro según los procedimientos establecidos en el Instituto de Biotecnología de las Plantas (IBP).
Cepa de hongo filamentoso
Se empleó micelio liofilizado de la cepa Mycosphaerella fijiensis Morelet CCIBP-Pf83 perteneciente a la Colección de Cultivos Microbianos del IBP.
Extracción de ADN
Para la extracción
de ADN se empleó el protocolo propuesto por Xin et al. (2003).
Se tomaron segmentos de 5 mm2 de tejido fresco de cada una de las
especies vegetales y del micelio liofilizado de M. fijiensis y se introdujeron
en tubos de 200 µl. Posteriormente, se añadieron 50 µl del
tampón A (100 mM NaOH, 2% Tween 20). Este tampón se prepara en
el momento de utilizarse a partir
de las soluciones de NaOH 10 M y Tween 20 al 20%. Las muestras fueron incubadas
durante 10 min a 95ºC y a continuación se añadieron
50 µl del tampón B (150 mM Tris-HCl pH 2, 2 mM EDTA), se mezclaron
moderadamente para homogenizar los tampones A y B y se tomaron 2 µl de
cada muestra para la reacción de PCR.
Condiciones de PCR
La reacción se realizó en un volumen total de 20 µl, que contenía 1X del tampón Dream Taq. (10X) (20 mM de MgCl2, 200 µM de la mezcla de dNTPs, 0.4 mM de los cebadores específicos para cada región del genoma analizado) (Tabla 1) y 1U de la enzima polimerasa Dream Taq (5U µl-1).Todos los ingredientes de la reacción provenían de Fermentas. Además, se incluyó en la reacción de PCR, 0.1% de BSA (m/v) y 1% de PVP 44 000 (m/v) para contrarrestar efectos inhibitorios en el PCR de diferentes compuestos comunes en los extractos crudos.
Las bandas obtenidas en el PCR se sometieron a una electroforesis en gel de agarosa al 1.5% y posteriormente fueron visualizadas en un transiluminador ultravioleta previa tinción en solución de bromuro de etidio.
RESULTADOS Y DISCUSIÓN
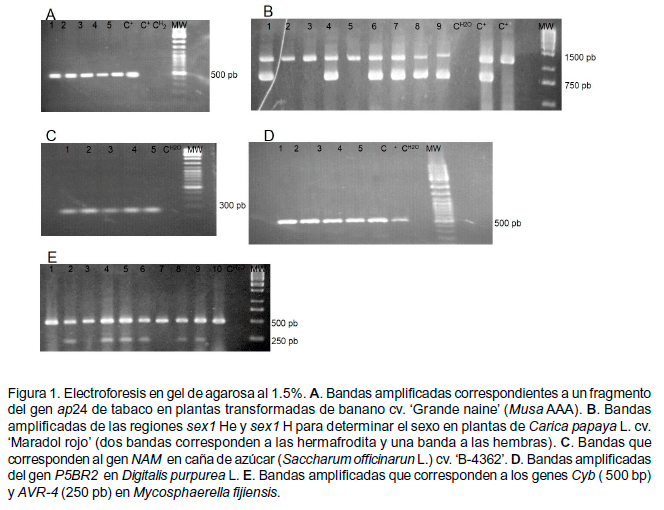
Mediante el método de extracción empleado fue posible amplificar las regiones de interés en el genoma de cada uno de los materiales vegetales analizados y la cepa de hongo filamentoso (Figura 1).
Con la adición
de 1% de PVP 44 000 (m/v) y 0.1% de BSA (m/v) se contrarrestaron los efectos
inhibitorios en la reacción de PCR. En experimentos previos (datos no
mostrados) estos compuestos no se utilizaron y la amplificación de las
secuencias diana no fue efectiva. Esto se debe en general a que en los extractos
crudos que se obtienen con los métodos de lisis alcalina existe una gran
variedad de compuestos contaminantes que inhiben la acción de la enzima
Taq polimerasa (Klimyuk et al., 1993). Posteriormente, al añadir
PVP y BSA en la reacción los resultados fueron los esperados (Figura
1). Estos resultados coinciden con los informados por Xin et al.
(2003) en otros cultivos. Es un método de extracción fácil
de ejecutar, reduce costos y permite monitorear regiones de interés en
el genoma de diferentes especies. Con este protocolo no se utilizan solventes
orgánicos que son dañinos para la salud, se prescinde del uso
de nitrógeno líquido,
de equipamiento costoso y es posible culminar la tarea de extracción
en 15 minutos por muestra.
CONCLUSIONES
Se demostró que mediante el protocolo propuesto por Xin et al. (2003) fue posible realizar extracciones de ADN genómico por lisis alcalina para el análisis por PCR de regiones de interés en cuatro especies vegetales y un hongo filamentoso.
REFERENCIAS
Klimyuk, VI, Carroll BJ, Thomas CM, Jones JD (1993) Alkali treatment for rapid preparation of plant material for reliable PCR analysis. Plant J. 3: 493-494
Ma Hao, Congfeng
Song, Wayne Borth, Diane Sether, Michael Melzer, John Hu (2011) Modified
expression of alternative oxidase in transgenic tomato and petunia affects the
level of tomato spotted wilt virus resistance. Biotechnology 11: 96
Pan Yong-Bao (2010)
Databasing Molecular Identities of Sugarcane (Saccharum spp.) Clones
Constructed with Microsatellite (SSR) DNA Markers. American Journal of Plant
Sciences 1: 87-94
Zhang Xin, Jeff
PV, Melvin JO, John JB (2003) High-Throughput DNA Extraction Method Suitable
for PCR. BioTechniques 34: 820-82
Recibido: 05-05-2014
Aceptado: 08-07-2014
Copyright (c) 2016 Biotecnología Vegetal
![]()
Biotecnología Vegetal eISSN 2074-8647, RNPS: 2154. ISSN 1609-1841, RNPS: 0397 Editada por: Instituto de Biotecnología de las Plantas. Universidad Central Marta Abreu de Las Villas. Carretera a Camajuaní km 5.5, Santa Clara, Villa Clara, Cuba CP 54 830 Tel: 53 42200124, e-mail: info@ibp.co.cu

Biotecnología Vegetal está bajo una Licencia Creative Commons Atribución-NoComercial 4.0 Internacional.