ARTÍCULO ORIGINAL
Comparación de tres métodos para la extracción de ARN total a partir de hojas de cacao
Comparison of three total RNA extraction methods from cacao leaves
Alinne A. Martínez-López*, Julia M. Lesher, Manuel E. Jiménez-García. *Autor para correspondencia.
División Académica de Ciencias Biológicas. Universidad Juárez Autónoma de Tabasco. Carretera Villahermosa-Cárdenas, kilómetro 0.5 entronque con Bosques de Saloya C.P. 86 039, Tabasco, México. e-mail: alinne_audrei@hotmail.com
RESUMEN
El establecimiento de un método de extracción de ARN total de plantas que cumpla con los criterios requeridos de pureza y rendimiento, es un paso crítico para estudios moleculares posteriores, sobre todo en plantas que son recalcitrantes y poseen de manera natural una alta cantidad de polifenoles, polisacáridos y proteínas. Estos se han referido como los principales contaminantes en los métodos de extracción. En este trabajo se compara la eficiencia de tres métodos de extracción de ARN de hojas de cacao (Theobroma cacao L.) para emplearse posteriormente en análisis moleculares. Se siguieron los protocolos establecidos y evaluó la cantidad y calidad del ARN obtenido. Fue posible reproducir los tres métodos, aunque se constataron diferencias respecto al tiempo de desarrollo y la calidad del ARN. El mejor método para la extracción de ARN total fue el de TRI-Reagent, el cual permitió obtener ARN con las características de calidad y concentración necesarias para futuros estudios, además de la facilidad de su empleo y el número de muestras que pueden ser ensayadas.
Palabras clave: laboratorios, polifenoles, protocolo.
ABSTRACT
The establishment of a method for extracting total RNA in plants that meets the required criteria of purity and yield is a critical step for subsequent molecular studies, especially in recalcitrant plants with naturally high amounts of polyphenols, polysaccharides and proteins. These have been referred to as the main contaminants in extraction methods. In this study the efficiency of three methods of RNA extraction from cacao leaves (Theobroma cacao L.) for later use in molecular analyzes were compared. Established protocols were followed and assessed the quantity and quality of RNA obtained. It was possible to reproduce the three methods, but differences were found regarding the development time and quality of RNA obtained. The best method for extraction of total RNA was TRI- Reagent, which allowed obtaining RNA with the characteristics of quality and concentration necessaries for future studies, in addition to ease of use and the number of samples that can be assayed.
Keywords: method, laboratory, polyphenols.
INTRODUCCIÓN
La extracción de ácido ribonucleico (ARN) total, es un procedimiento de rutina en los laboratorios de biología molecular y es la base en estudios que involucran la expresión y búsqueda de genes. A la fecha, se han desarrollado numerosos métodos, además de kit de reactivos que son ofrecidos por las distintas casas comerciales con este objetivo. Ocasionalmente en los laboratorios de investigación, la reproducibilidad de alguno, se ve limitada por la diferencia entre marcas de equipos, reactivos, así como por el número de muestras que pueden ser ensayadas durante la fase de prueba para el tejido de interés.
Teniendo en cuenta, que el aislamiento del ARN de los metabolitos mencionados es el principal interés del uso de los métodos de extracción, se planteó como objetivo de este trabajo comparar la eficiencia de tres métodos de extracción de ARN para emplearse posteriormente en análisis moleculares. El primero de ellos se basa en el desarrollado por Chomczynski y Sacchi (1987 y 2006) y su nombre comercial es TRI-Reagent (Sigma) y dos métodos utilizados para especies recalcitrantes: el referido por Bekesiova et al. (1999) para la planta carnívora Drosera rotundifolia y el descrito por da Silva et al. (2003), específicamente para cacao, el cual se basa en precipitaciones diferenciales con terbutanol.
Particularmente, cuando se trabaja con especies recalcitrantes, se hace necesaria la estandarización del método de extracción de entre las opciones existentes, para lograr un rendimiento y pureza adecuada del ARN total obtenido. Esta etapa se considera determinante (Jones et al., 1997; Tattersall et al., 2005; Rodríguez-Ávila et al., 2009).
El cacao (Theobroma cacao L.) se considera una especie recalcitrante para la extracción de ácidos nucleicos, debido a que contiene una alta cantidad de polifenoles y polisacáridos en los diferentes tejidos, los cuales co-precipitan junto con los ácidos nucleicos (da Silva et al., 2003; Quiñones et al., 2013).
MATERIALES Y MÉTODOS
Material vegetal
Se colectaron hojas de cinco plantas de cacao del grupo genético Forastero, las cuales fueron muestreadas en tres plantaciones diferentes establecidas en el estado de Tabasco, México. Las hojas fueron desinfectadas externamente con una solución de hipoclorito de sodio al 0.5%, se lavaron con agua destilada para eliminar el cloro residual y se trituraron con nitrógeno líquido hasta convertirlas en un polvo fino que se almacenó a -80ºC. Para la evaluación de los protocolos de extracción se tomaron 50 o 100 mg de tejido vegetal, con una extracción única por individuo.
Preparación de materiales para extracción de ARN
La cristalería utilizada fue sumergida en una solución de NaOH 1N y esterilizada durante 4 h a 120°C con 1 atm de presión y los materiales de plástico se desinfectaron con etanol absoluto y fueron esterilizados por 45 min durante 20 min a 120°C con 1 atm de presión. Todas las soluciones que lo requerían fueron preparadas en agua tratada con dietil pirocarbonato (DEPC) al 0.1%, a menos que el protocolo indicara lo contrario.
Métodos de extracción de ARN utilizados
- Método de TRI-Reagent
Se utilizó el protocolo proporcionado por la casa comercial Sigma-Aldrich (Chomczynski y Sacchi 1987 y 2006) que se describe brevemente a continuación:
Se adicionaron 200 µl del TRI-Reagent al tejido, el cual se colocó previamente en tubos (Eppendorf) de 2 ml, la muestra se homogenizó con un micropistilo y se dejó a temperatura ambiente (TA) por 24 h. Después de ese periodo se completó el volumen de reactivo en cada muestra a 1 ml, se aplicó el apartado `a' de notas, en la sección de Preparación de la muestra del protocolo para separar las partes fibrosas del lisado por centrifugación de las muestras a 12 000 g durante 10 min a 4°C y se transfirió la fase clara del sobrenadante a un tubo limpio de 2 ml donde se adicionaron 0.2 ml de cloroformo. Las muestras se agitaron vigorosamente por 15 s por inversión del tubo y posteriormente se dejaron a TA por 15 min. Posteriormente, las muestras se centrifugaron a 12 000 g durante 15 min a 4°C con lo cual se obtuvieron tres fases: una fase roja orgánica, la interfase y la fase superior acuosa, la que se transfirió cuidadosamente a un tubo limpio y se le adicionaron 0.5 ml de isopropanol. Para precipitar el ARN, las muestras se dejaron 10 min a TA e inmediatamente se centrifugaron a 12 000 g por 10 min a 4°C, el sobrenadante fue descartado. El ARN precipitado, se lavó dos veces con 1 ml de etanol al 75% (v/v) mediante agitación con vortex y se recolectó al fondo del tubo por centrifugación durante 5 min a 7 500 g y 4°C. El precipitado se dejó secar en estufa a 37°C hasta obtener la completa evaporación del etanol y finalmente se resuspendió en un volumen adecuado de agua tratada con DEPC 0.1%. Las muestras se almacenaron a -70 °C.
- Método CTAB modificado por Bekesiova et al. (1999)
El tampón de extracción se preparó con 2% CTAB (m/v), 100 mM Tris-HCl (pH 8.0), 25 mM EDTA sódico (pH 8.0), 2 M NaCl y se adicionó â-mercaptoetanol al 2% justo antes de utilizarlo. Para la extracción de ARN se precalentaron 700 µl de tampón de extracción a 65°C y el tejido triturado previamente, se transfirió al tubo que contenía el tampón y se agitó vigorosamente. La extracción del lisado se realizó dos veces con volúmenes iguales de cloroformo alcohol: alcohol isoamílico 24:1. Posteriormente, las muestras se centrifugaron a 10 000 rpm por 10 min a 4°C y a la fase acuosa obtenida se le agregó 0. 25 vol. de LiCl~10 M y se mezcló bien por pipeteo. Para precipitar el ARN, las muestras se incubaron toda la noche a 4°C. El precipitado se obtuvo centrifugando las muestras a 10 000 rpm por 20 min a 4°C y se resuspendió en agua tratada con DEPC. Para precipitar nuevamente el ARN, se adicionó 0.1 vol de Acetato de Sodio (pH 5.2) y 2.5 vol de etanol al 96% e incubación a -70°C por más de 30 min. Las muestras se centrifugaron a 10 000 rpm por 20 min a 4°C para obtener el precipitado, el cual se lavó con etanol al 70% y se resuspendió en agua tratada con DEPC. Las muestras se almacenaron a -70°C.
- Método de Terbutanol referido por da Silva et al. (2003)
Alícuotas
del tejido se colocaron en tubos de 2 ml y se adicionó 1 ml de tampón
de homogenización (0.2 M Ácido Bórico, 10 mM de EDTA pH
7.6 ajustado con Tris), 20 µl SDS al 25% y 20 µl de â-mercaptoetanol.
Las muestras se agitaron por 10 min en el equipo Eppendorf MixMate®
a 300 rpm TA y se centrifugaron a 15 000 g por 10 min a TA. La fase acuosa se
transfirió a un tubo nuevo e inmediatamente se adicionó 1 ml de
fenol/cloroformo/alcohol isoamílico (25:24:1). Se repitió la adición
de tampón, agitación y centrifugación y la fase acuosa
obtenida se transfirió a un tubo nuevo, anotando el volumen obtenido.
Seguidamente, se adicionó 1 ml de cloroformo/ alcohol isoamílico
(24:1) y se realizó la primera separación de los ácidos
nucleicos de los polisacáridos. Para ello se adicionaron 0.1 vol. de
Acetato de Sodio pH 4.5 más 4.5 vol. de terbutanol, tomando en cuenta
el volumen obtenido. Los tubos se colocaron en hielo y agitación usando
el Eppendorf MixMate ® a 300 rpm. Luego las muestras se centrifugaron a
15 000 g por 10 min a 4°C y el sobrenadante se recuperó cuidadosamente
en un tubo nuevo y se mezcló con 0.4 vol. de terbutanol. Los tubos se
colocaron en hielo durante 30 min e inmediatamente las muestras se centrifugaron
a 15 000 g por 20 min a 4°C. El precipitado obtenido se lavó con
etanol al 70% y se resuspendió en 900 µl de tampón de homogenización.
Para la segunda precipitación diferencial de polisacáridos, se
aplicaron los mismos pasos de la primera separación tomando en cuenta
como volumen inicial el utilizado del tampón de
homogenización. El precipitado final se secó en estufa a 37°C
y se resuspendió en agua tratada con DEPC. Las muestras se almacenaron
a -70°C.
Tratamiento de las muestras con DNasa
Para remover la contaminación por ADN, en todos los métodos, se utilizó la enzima DNase I siguiendo el protocolo indicado por el fabricante (BioBasic). Una vez tratadas, se eliminó la enzima de la muestra por la adición de 1 ml de fenol/cloroformo/alcohol isoamílico (25:24:1). La mezcla se agitó por 10 min y posteriormente se centrifugó a 15 000 g por 10 minutos a TA. El sobrenadante fue transferido a un tubo limpio. La precipitación del ARN se hizo con 0.1 vol. de Acetato de sodio 3 M y 2.5 vol. de etanol absoluto y se dejó a TA por 15 min. El precipitado se obtuvo al centrifugar la muestra a 12 000 g por 10 min a 4°C llevando a cabo un lavado con etanol al 70%.
Evaluación de la cantidad y la calidad del ARN obtenido
La pureza del ARN fue estimada por la medida del radio de absorbancia a A260/A280 con un espectrofotómetro SmartSpec TM Plus BIO-RAD. Las muestras fueron visualizadas en gel desnaturalizante de agarosa-formaldehído al 1%, utilizando como tampón de electroforesis MOPS 1X (Bio-Rad). Previamente las muestras se desnaturalizaron colocando en tubos de 0.5 ml aproximadamente ~1 µg de ARN total, 2 µl de Agua tratada con DEPC, 5 µl de formamida, 2 µl de formaldehído al 37%, 1 µl MOPS 10X. Por último, las muestras se calentaron a 65°C por 5 min y se colocaron en hielo. El Azul de bromofenol se utilizó como indicador de frente de corrida. La tinción se realizó con Bromuro de Etidio 1mg ml-1. Los geles fueron analizados en un trasluminador de luz UV uvitec.
RESULTADOS Y DISCUSIÓN
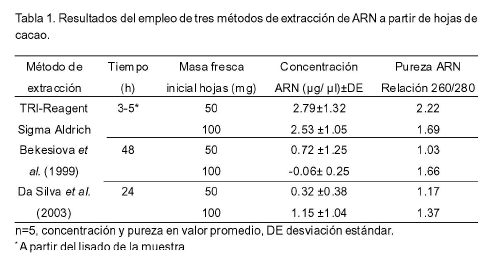
Mediante los protocolos descritos fue posible reproducir los tres métodos evaluados para la extracción de ARN de hojas de cacao, aunque se constataron diferencias respecto al tiempo de desarrollo y la calidad del ARN obtenido (Tabla 1).
A pesar de
que los autores refieren que cada método proporciona facilidad y ahorro
de tiempo durante su realización, en la práctica variaron considerablemente.
Por ejemplo, en el método de TRI-Reagent en la parte de homogenización,
al ir aumentando el tiempo después de la adición del reactivo, el
tejido se observó de una coloración café y al finalizar las
24 h, el lisado era homogéneo. Sin embargo, el precipitado obtenido fue
de una coloración oscura, incluso algunos tuvieron una consistencia arenosa
de difícil solubilización.
En los primeros pasos de separación del protocolo descrito por Bekesiova et al. (1999) el lisado se observó de una coloración verdosa, probablemente por la presencia de pigmentos del tipo de clorofilas, los cuales al separarse las fases se encuentran principalmente en la parte inferior. Además, el precipitado no fue visible durante la precipitación final con Acetato de Sodio sino hasta el posterior tratamiento con DNasa. En contraste, en el protocolo propuesto por da Silva et al. (2003) la acción mecánica de la agitación prolongada en los diferentes pasos, resultó en la lisis exitosa del tejido y en la eliminación de los metabolitos conforme se van utilizando las diferentes mezclas de solventes. Con el empleo de este protocolo el precipitado fue de color blanco y muy pequeño.
Se ha informado por otros autores que cuando los polifenoles son liberados durante la lisis celular, disminuye la calidad y el rendimiento de la extracción de ácidos nucleicos, debido a que mediante la enzima polifenoloxidasa con su actividad catecolasa los polifenoles se oxidan a quinonas y se acoplan covalentemente a los ácidos nucleicos degradándolos o provocando que precipiten con ellos (Loomis, 1974; Salzman et al., 1999; Rojas et al., 2011). Esto pudiera explicar la coloración oscura de todos los precipitados obtenidos en el método de TRI- Reagent.
Respecto a los problemas que se presentaron en el desarrollo de los métodos, el protocolo descrito por da Silva et al. (2003), aunque no fue en el que se consumió mayor tiempo, representó un mayor desgaste físico para el analista. En primer lugar, por requerir un mayor número de pasos y en segundo, por la pérdida de muestras o tiempo debido al uso del terbutanol, el cual es altamente higroscópico y requiere de una temperatura cálida para mantener su estado líquido, contrario a las temperaturas utilizadas en el protocolo. Esto resultaba en ocasiones en la cristalización del reactivo dentro de las puntas de micropipeta y de las muestras.
Tanto en el protocolo de Bekesiova et al. (1999), como en el de da Silva et al. (2003), se observó la presencia de películas aceitosas y mucosas en la interfase en los pasos de separación, en ocasiones tan poco densas que eran llevadas junto al sobrenadante al ser transferidas a un tubo limpio. Cuando lo anterior sucedía, se observó que las proteínas ocupaban un volumen importante, dejando minimizada la fase acuosa.
El rendimiento promedio del ARN total obtenido fue de aproximadamente 2.5 µg únicamente para el protocolo de TRI-Reagent sin diferencias con respecto a las cantidades iniciales de tejido evaluadas, mientras que la concentración obtenida con los protocolos restantes fue menor. Además, se observó diferencia entre la cantidad utilizada de material vegetal de inicio, pues para Bekesiova et al. (1999) una menor cantidad representó una mayor concentración, los valores en algunos casos fueron negativos, no así en da Silva et al. (2003) donde a mayor cantidad de tejido, hubo una mayor concentración (Tabla 1). A pesar de que los valores de concentración aún son bajos en los protocolos evaluados, el tratamiento con DNasa aumentó la pureza de la muestra, pues anterior a ello la mayoría de estos valores resultaron negativos (datos no mostrados).
La calidad del ARN obtenido del tejido de hojas de cacao fue adecuada. El protocolo de TRI- Reagent presentó el mejor valor en cuanto a pureza de acuerdo con el radio A260/A280 (2.2). En los dos protocolos restantes, los valores fueron menores.
La electroforesis permitió la visualización de la banda ribosomal 28 S (Figura 1). Los valores anteriores tanto de rendimiento como de calidad del ARN obtenido confirman que a pesar de los diferentes pasos de separación de los materiales orgánicos y contaminantes, las muestras finales todavía presentaron remanentes de ciertos compuestos que interfirieron en su calidad. Sin embargo, la visualización de las bandas también confirma que los diferentes reactivos utilizados para neutralizar las ribonucleasas presentes en los tampones utilizados, como el â-mercaptoetanol y el tiocianato de guanidina, fueron suficientes para mantener un ARN íntegro aunque con la presencia de ciertas proteínas de difícil separación (Rojas et al., 2011).
En las plantas, es común la formación de estructuras de protección, las cuales poseen paredes celulares reforzadas con capas de celulosa, lignina y otros compuestos fenólicos. También se refiere la presencia de enzimas como la fenilalanina amonialasa, peroxidasa y polifenoloxidasa (Simo et al., 2011). Estas características como menciona Rojas et al.(2011), complejizan mucho más el escenario molecular en las plantas recalcitrantes como el cacao, debido a que los compuestos del tipo de fenoles y polisacáridos, se encuentran dentro de los principales contaminantes en las extracciones de ARN, al evitar su solubilización después de la precipitación con alcoholes (Sambrook y Russell, 2001).
Es muy conocido
el gran número de métodos originales y modificados, que en su
mayoría informan modestos rendimientos y ARN parcialmente intactos (Wang
et al., 2008; Wang et al., 2011), los cuales no pueden ser generalizados
o aplicados a distintas especies sobre todo para extracción de ARN, donde
el contenido de este ácido nucleico es relativo al momento y estado de
las células a evaluar. Metodologías confiables y probadas, en
ocasiones no pueden ser implementadas en todos los laboratorios, por el costo
de ciertos reactivos o la dificultad para su adquisición en el laboratorio
de trabajo. Aunque los resultados promedios de los protocolos evaluados difieren,
sí se obtuvieron muestras que cumplieron con las características
de rendimiento y calidad, las cuales pueden ser utilizadas en la aplicación
de técnicas moleculares más avanzadas.

CONCLUSIONES
La extracción de ARN total requiere de la aplicación de metodologías que en conjunto optimicen los rendimientos de obtención en combinación con bajos costos económicos y de tiempo. Los tres métodos evaluados fueron replicados exitosamente en el laboratorio. El mejor método para la extracción de ARN total fue el de TRI-Reagent, el cual permitió obtener muestras de ARN con las características de calidad y concentración necesarias para futuros estudios, además de la facilidad de su empleo y el número de muestras que se pueden ser ensayadas. Los métodos restantes también resultaron efectivos, sin embargo, el tiempo para su realización y la cantidad de consumibles empleados hizo que fueran las últimas opciones para ser establecidos en laboratorio.
AGRADECIMIENTOS
Los autores agradecen a la familia Walter de la hacienda La Luz, a la profesora Carmina y el Sr. Juan López por su valioso apoyo en la colecta del material vegetativo, así como a los profesores que apoyaron con el transporte.
REFERENCIAS
Bekesiova, I, Nap JP, Mlynarova L (1999) Isolation of high quality DNA and RNA from leaves of the carnivorous plant Drosera rotundifolia. Plant Molecular Biology Reporter 17: 296-277
Chomczynski, P, Sacchi N (1987) Single-step method of RNA isolation by acid guanidinium thiocyanate-phenolchloroform extraction. Anal Biochem 162: 156_159
Chomczynski, P, Sacchi N (2006) The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: twenty-something years on. Nat Protoc 1:581_585
da Silva, AG, Micheli F, Fortes C, de Mattos JC (2003) Isolation and purification of functional total RNA from different organs of cacao tree during its interaction with the pathogen Crinipellis perniciosa. BioTechniques 35:494-500
Jones, CS, Iannetta PPM, Woodhead M, Davies HV, McNicol RJ, Taylor MA (1997) The isolation of RNA from raspberry (Rubus idaeus) fruit. Molec Biotech 8: 219_221
Loomis, WD (1974)
Overcoming problems of phenolics and quinones in the isolation of plant
enzymes and organelles. Methods in Enzymology 31: 528-545
Quiñones, J, Trujillo R, Capdesuñer Y, Quirós Y, Hernández, M (2013) Potencial de actividad antioxidante de extractos fenólicos de Theobroma cacao L. (cocoa). Revista cubana de plantas medicinales 18 (2): 201-2015
Rodríguez-Ávila, NL, Narváez-Zapata JA, Aguilar-Espinoza ML, Rivera-Madrid R (2009) Full-Length gene enrichment by using an optimized RNA Isolation Protocol in Bixa orellana recalcitrant tissues. Mol Biotechnol 42:84-90
Rojas, L, Portal O, Jiménez E (2011) Extracción de ARN total en plantas y hongos filamentosos. Biotecnología Vegetal 11 (4): 213 _ 222
Salzman, RA, Fujita T, Zhu-Salzman K, Hasegawa PM, Bressan RA (1999) An improved RNA isolation method for plant tissues containing high levels of phenolic compounds or carbohydrates. Plant Molecular Biology Reporter 17: 11-17
Simo, C, Djocgoue PF, Mbouobda HD, EFFA PO, Boudjeko T, Omokolo DN (2011) Variation and heredability of polyphenoloxidasic activities in two hybrid families of Theobroma cacao L. after cocoa pods inoculation with Phytophtora magakarya Bras. et Grif. Plant Pathology Journal 10(3): 89-98
Sambrook, J, Russell DW (2001) Molecular cloning: a laboratory manual. Third edition. Cold Spring Harbor Laboratory Press
Tattersall, EAR, Ergul A, AlKayal F, DeLuc L, Cushman JC, Crame, GR (2005) Comparison of Methods for Isolating High-Quality RNA from leaves of grapevine. Am. J. Enol. Vitic. 56: (4) 400-407
Wang, P, Qi M, Barboza P, Beth M, Ungerfeld E, Brent L, McAllister TA, Forster RJ (2011) Isolation of high-quality total RNA from rumen anaerobic bacteria and fungi, and subsequent detection of glycoside hydrolases. Can. J. Microbiol. 57: 590_598
Wang, X, Tian W, Li Y (2008) Development of an efficient protocol of RNA isolation from recalcitrant tree tissues. Molecular Biotechnology 38: 57-64
Wang, X , Wang B, Liu L, Cui X, Yang J, Wang H , Jiang H, Luo B, Long Z , Dou W, Zhang N, Peng D (2010) Isolation of high quality RNA and construction of a suppression subtractive hybridization library from ramie (Boehmeria nivea L. Gaud.) Mol Biol Rep 37:2099_2103
Recibido: 1-3-2013
Aceptado: 10-4-2013
Copyright (c) 2016 Biotecnología Vegetal
![]()
Biotecnología Vegetal eISSN 2074-8647, RNPS: 2154. ISSN 1609-1841, RNPS: 0397 Editada por: Instituto de Biotecnología de las Plantas. Universidad Central Marta Abreu de Las Villas. Carretera a Camajuaní km 5.5, Santa Clara, Villa Clara, Cuba CP 54 830 Tel: 53 42200124, e-mail: info@ibp.co.cu

Biotecnología Vegetal está bajo una Licencia Creative Commons Atribución-NoComercial 4.0 Internacional.
