Artículo original
Biotecnología Vegetal Vol. 17, No. 2: 99 - 104, abril - junio, 2017
Instituto de Biotecnología de las Plantas. UCLV. MES.
eISSN 2074-8647, RNPS: 2154
Determinación de la estabilidad genética de plantas propagadas in vitro de Vasconcellea x helbornii Badillo (Badillo) con el uso de marcadores ISSR
Determination of genetic stability of in vitro propagated plants of Vasconcellea x helbornii Badillo (Badillo) using ISSR markers
Mónica Jadán1,2, Shirley Brito1, Claudia Segovia1, Rafael Gómez-Kosky2, Idalmis Bermúdez-Caraballoso2, Karina Proaño1
1 Departamento de Ciencias de la Vida, Universidad de las Fuerzas Armadas ESPE. Ave Gral Rumiñahui. Sangolquí. Quito. Ecuador. PO BOX 171-5-31B. e-mail: mbjadan@espe.edu.ec; monica@ibp.co.cu
2 Instituto de Biotecnología de las Plantas, Universidad Central Marta Abreu de Las Villas. Carretera a Camajuaní km 5,5. Santa Clara. Villa Clara. Cuba. CP 54 830.
RESUMEN
Los papayuelos de montaña, entre ellos el babaco [Vasconcellea x helbornii Badillo (Badillo)], son originarios del Ecuador y poseen un alto valor nutricional por lo que tiene gran demanda a nivel nacional e internacional. El presente trabajo tuvo como objetivo determinar a través de marcadores ISSR la estabilidad genética de plantas de babaco propagadas in vitro. Se utilizaron hojas jóvenes (uno y dos) de 10 plantas madre (estacas) y de 20 plantas propagadas in vitro. Para el análisis se emplearon ocho cebadores. Un total de 55 bandas fueron observadas de las 30 plantas analizadas. Se observó un 100% de loci monomórficos entre las plantas madre provenientes de estacas y las plantas propagadas in vitro. Este análisis indicó que no existieron diferencias a nivel molecular entre las plantas analizadas, para las combinaciones de cebadores empleadas y que los marcadores ISSR pueden ser usados para este propósito.
Palabras clave: babaco, marcadores moleculares, organogénesis, variación somaclonal
Abstract
The mountain papayuelo, among them babaco [Vasconcellea x helbornii Badillo (Badillo)], are native of Ecuador and has a great nutritional value, that is it has a huge national and international demand. The objective of the present work was to determine the genetic stability of in vitro propagated plants of babaco using ISSR markers. It were used young leaves (one and two) of 10 mother plants (cuttings) and from 20 plants in vitro propagated. For the analysis, eight primers were used. Forty five bands were observed from the 30 plants analyzed. It were observed 100% of monomorphic loci between stem plants and plants in vitro propagated. This analysis indicates that there were no differences at the molecular level between the plants analyzed for the combinations of primers used and the ISSR markers may use for this purposes.
Keywords: babaco, molecular markers, organogenesis, somaclonal variation
INTRODUCCIÓN
Las especies del género Vasconcellea se cultivan en zonas altas de Sur América especialmente en Ecuador, Colombia y Perú (Schelderman et al., 2011). De las 16 especies que posee Ecuador, el babaco al ser una planta nativa del país muestra un gran potencial como especie cultivable (Robles et al., 2013).
Durante generaciones la propagación de babaco se ha realizado mediante el uso de estacas, el cual es un sistema poco confiable de producción de plantas a gran escala, por su alto costo y por la propagación de enfermedades. Las técnicas de cultivo in vitro son muy útiles para la obtención de plantas de alta calidad genética y fitosanitaria, donde los métodos tradicionales son difíciles de aplicar. El establecimiento de un protocolo eficiente de regeneración de plantas a través de técnicas de cultivo de tejidos en Vasconcellea permite la multiplicación de nuevos individuos, conservación de germoplasma y producción de semillas de mejores características agronómicas (Vélez-Mora et al., 2015).
Sin embargo, las plantas regeneradas por cultivo de tejidos son propensas a alteraciones genéticas debido al estrés inducido por las condiciones de cultivo in vitro y el modo de regeneración. La estabilidad genética constituye un requisito esencial para su uso (Bhowmik et al., 2016).
En este sentido, se ha observado variabilidad genética en especies como: Actinidia deliciosa (A. Chev.) C.F. Liang & A.R. Ferguson (Palombi y Damiano, 2002), Malus pumila L. (Mogdil et al., 2005) y en Vanilla planifolia Jacks, propagadas vía organogénesis indirecta, donde obtuvieron un 71.66% de polimorfismo (Ramírez y Iglesias, 2015). La variabilidad genética ha estado asociada con el genotipo, el tipo de explante, la composición del medio de cultivo, la forma de regeneración, entre otros factores (Yin et al., 2013).
En babaco se han desarrollado varios trabajos relacionados con el cultivo in vitro (Vega y Kitto, 1991; Jordán y Piwanski, 1997; Jordán y Piwanski, 1998; Jordán et al., 2009; Jadán et al., 2016 b) pero no se han realizado estudios referidos a la estabilidad genética de las plantas propagadas.
Los marcadores moleculares para determinar la estabilidad genética de plantas regeneradas in vitro, han sido ampliamente utilizados. Los ISSR (del inglés: Inter Simple Sequence Repeats) presentan una alta reproducibilidad (Rout et al., 2009; Mohanty et al., 2011; Nayak et al., 2011; Parida et al., 2011; Leva y Petruccelli, 2012). Ellos se basan en el uso de secuencias de microsatélites como cebadores que generan múltiples bandas relacionadas. Varios marcadores tipo microsatélites permiten analizar un gran número de loci, lo que da una alta posibilidad de encontrar polimorfismo, incluso entre genotipos relacionados (Carrasco et al., 2009). Son marcadores simples y rápidos que combinan la mayoría de las ventajas de otros tipos de marcadores tales como: SSR (del inglés: Simple sequence repeats), AFLP (del inglés: Amplified fragment length polymorphism), RAPD (del inglés: Random amplified polymorphic DNA) (Ramakrishnan et al., 2014) entre otros.
En babaco, los marcadores ISSR, solo se han empleado para caracterizar la diversidad genética y el flujo de genes entre poblaciones (Kyndt et al., 2005). Sin embargo, atendiendo a sus ventajas podrían utilizarse en estudios de análisis de la estabilidad genética.
Por todo lo antes expuesto, el presente trabajo tuvo como objetivo determinar a través de marcadores ISSR la estabilidad genética de plantas de babaco propagadas in vitro.
MATERIALES Y MÉTODOS
Material vegetal
Muestras de hojas jóvenes (uno y dos) de 10 plantas madre de babaco (estacas) (PM1-PM10) obtenidas de acuerdo con el protocolo de Jadán et al. (2016 a) y hojas jóvenes de plantas obtenidas in vitro (Jadán et al., 2016 b) con dos meses de cultivo en la fase de aclimatización. Se emplearon dos repeticiones por cada una (1-20).
Determinación de la estabilidad genética mediante ISSR
Se tomaron 200 mg de muestra foliar y se trituraron con nitrógeno líquido hasta formar un polvo fino, de acuerdo con el protocolo de Khanuja et al. (1999) modificado. Una vez extraído el ADN se procedió a preparar el gel de agarosa al 1.0% para determinar su integridad. Posteriormente, se cuantificó y verificó la pureza por espectrofotometría a través de las relaciones 260/280, 260/230 mediante Nanodrop 2000 (Thermo Scientific, EE. UU).
Se emplearon ocho cebadores (Ochoa et al., 2008) que se describen en la Tabla 1.

La amplificación por reacción en cadena de la polimerasa (PCR) se llevó a cabo en 25 µl de volumen final, que incluyó 10 ng de la mezcla de ADN genómico, 2.5 μl de tampón 10xPCR, 50mM de Cloruro de Magnesio (MgCl2), 10 mM de dNTP, 2 µl del cebador, 0.4 U de Taq Polimerasa (5 U/µl) (Invitrogen®), y 18.2 μl de agua ultra-pura. El programa para la PCR fue: desnaturalización a 94°C durante 2 min, seguido por 35 ciclos bajo las siguientes condiciones: desnaturalización a 94°C por 30 s, hibridación por 45 s y extensión a 72°C por 2 min. Se realizó una extensión final a 72°C durante 7 min. Las reacciones de la PCR se llevaron a cabo en el termociclador TECHNE-TC-512 (EE.UU).
Una vez que se realizó la PCR se procedió al análisis de los amplicones, estos fueron visualizados en geles de agarosa al 1.5% (Dong et al., 2007). La corrida electroforética se realizó a 120 V, 300 mA durante 50 min y el gel fue analizado con el programa Dolphin-View Imagine System.
Todas las reacciones con cada cebador ISSR fueron repetidas dos veces. Se calculó el número total de bandas, el promedio de bandas por cebador y el porcentaje de loci monomórficos. Las bandas amplificadas se registraron en una matriz binaria de uno (presencia) y cero (ausencia). Se usó el coeficiente de similitud de DICE para el análisis entre las plantas madre y las obtenidas in vitro, con la ayuda del paquete estadístico GenAlEx 6 (Versión 6) (Peakall y Smouse, 2006) para análisis genéticos en Excel sobre Windows.
RESULTADOS Y DISCUSIÓN
A través del análisis ISSR se determinó la estabilidad genética de las plantas de babaco propagadas in vitro. Los ocho cebadores empleados produjeron bandas fuertes y claras. El número de bandas por cada cebador varió de 1-10 con un promedio de 6.8 bandas por cada uno. El cebador 17899A produjo el mayor número de bandas (10), con tamaño entre 350-2000 pares de bases. Se obtuvo un total de 55 bandas en las plantas analizadas (Tabla 2). Se observó un 100% de loci monomórficos entre las plantas madre provenientes de estacas y las plantas propagadas in vitro (Tabla 2 y Figura 1).
El coeficiente de similitud alcanzó el valor de uno entre las plantas madre de estacas y las propagadas in vitro lo que indicó 100% de similitud entre ellas. Este análisis permitió considerar que no existieron diferencias a nivel molecular para los cebadores analizados.

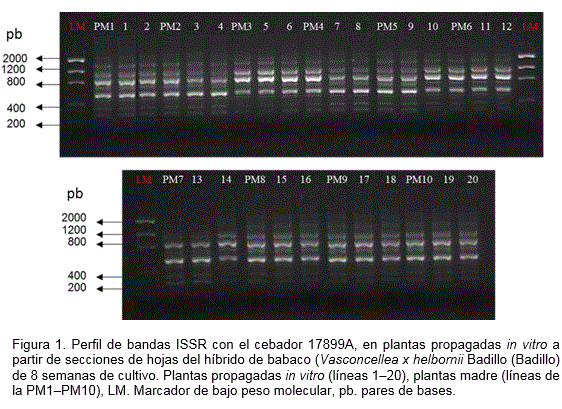
Todos los cebadores empleados en la presente investigación amplificaron las muestras de ADN analizadas. Ochoa et al. (2008) con estos mismos cebadores, en estudios de diversidad genética entre poblaciones de Polylepis incana Kunth y P. pauta Hieron, lograron la amplificación de 211 bandas de ambas poblaciones, de ellas 202 fueron polimórficas, lo que representó un 95.7% del total, con un promedio de 21 bandas por cebador. Ello demostró la amplia diversidad genética de ambas poblaciones. Sin embargo, para babaco con el empleo de estos cebadores no se encontró polimorfismo entre las plantas propagadas in vitro y las obtenidas de estacas, lo que demostró la efectividad de este tipo de marcador también para estudios de estabilidad genética.
Hasta el momento en Vasconcellea, con el empleo de marcadores ISSR, solo se han referido los trabajos desarrollados por Kyndt et al. (2005) para caracterizar la diversidad genética y flujo de genes entre diferentes poblaciones de babaco; Ocampo et al. (2006) y Kanupriya et al. (2012) para analizar el flujo de genes entre Carica papaya L. y especies de Vasconcellea y Carrasco et al. (2014) para caracterizar genéticamente la especie V. chilensis (Planch. ex A.DC.). Estos resultados constituyen la primera investigación relacionada con estudios de estabilidad genética en plantas de babaco obtenidas por cultivo in vitro a través de marcadores ISSR.
La estabilidad genética de plantas regeneradas a través de las técnicas de cultivo in vitro, ha sido referida en un gran número de especies, tales como: Prunus dulcis L. (Martins et al., 2004), Phoenix dactylifera L. (Saker et al., 2006), Jatropha curcas L. (Sharma et al., 2011), Cleome gynandra L. (Rathore et al., 2014), entre otras.
Yin et al. (2013), en Lilium spp. emplearon la misma vía de propagación de plantas a través de la organogénesis directa a partir de secciones de hojas y no detectaron variabilidad genética con el empleo de marcadores ISSR.
El empleo de estos marcadores en el presente estudio constituye un punto de partida importante para su uso en estudios de estabilidad genética de plantas obtenidas a través del cultivo in vitro para cualquiera de las especies de Vasconcellea.
CONCLUSIONES
Los marcadores ISSR pueden ser utilizados en estudios para determinar la estabilidad genética de plantas propagadas in vitro de babaco, con estos no se encontraron diferencias a nivel molecular entre las plantas in vitro y las plantas madre.
AGRADECIMIENTOS
Al Laboratorio de Biología Molecular de Plantas de la Universidad de las Fuerzas Armadas ESPE, Quito, Ecuador.
REFERENCIAS
Bhowmik S, Basu A, Sahoo L (2016) Direct Shoot Organogenesis from Rhizones of Medicinal Zingiber Alpinia calcarata Rosc and Evaluation of Genetic Stability by RAPD and ISSR Markers. Crop Sci Biotech 19(2): 157-165; doi:10.1007/s12892-015-0119-4
Carrasco B, Ávila P, Pérez-Díaz J, Muñoz P, García R, Díaz C, Lavandero B, Zurita A, Retamales J, Caligari P (2009) Genetic structure of Highland papayas (Vasconcellea pubescens (A.DC.) Badillo) cultivated along a geographic gradient in Chile as revealed by inter simple sequence repeats (ISSR). Genet Resour Crop Evol 56(3): 331-337; doi:10.1007/s10722-008-9367-1
Carrasco B, García R, Díaz C, Ávila P, Cáceres P, Lobos G, Silva H, Caligari D (2014) Genetic and morphological characterization of the endangered Austral papaya Vasconcellea chilensis (Planch, ex A. DC.) Solms. Genet Resour Crop Evol 61(7): 1423-1432; doi:10.1007/s10722-014-0143-0
Dong Y, Burch-Smith TM, Liu Y, Mamillapalli P, Dinesh-Kumar SP (2007) A ligation-independent cloning tobacco rattle virus vector for high-throughput virus-induced gene silencing identifies roles for NbMADS4-1 and -2 in floral development. Plant Physiol 145(4): 1161–1170; doi:10.1104/pp.107.107391
Jadán MG, Gómez-Kosky R, Bermúdez-Caraballoso I (2016 a) Obtención de plantas madre de Vasconcellea x helbornii (Badillo) Badillo a partir de estacas en condiciones semicontroladas. Biotecnología Vegetal 16(1): 13-20
Jadán MG, Basantez K, Gómez-Kosky R, Bermúdez-Caraballoso I (2016b) Establecimiento in vitro de brotes de Vasconcellea x helbornii (Badillo) Badillo. Biotecnología Vegetal 16(2): 67-72
Jordán M, Piwanski D (1997) Regeneration of babaco [Carica pentagona (Heilborn) Badillo] using leaf explants and shoot-tip culture. Rev Intl Bot Exp 61: 109-115
Jordán M, Piwanski D (1998) Regeneration of babaco [Carica pentagona (Heilborn) Badillo] by somatic embryogenesis. Revista Internacional de Botánica Experimental 64: 101-105
Jordán M, Vélez D, Armijos R (2009) Biotecnologías aplicables al desarrollo de algunas especies de Caricaceas cultivadas en la región Andina: avances y problemas. Revista Colombiana de Ciencias Hortícolas 3(1): 1-17
Khanuja PS, Ajit K, Darokar S, Kumar S (1999) Rapid Isolation of DNA from Dry and Fresh Samples of Plants Producing Large Amounts of Secondary Metabolites and Essential Oils. Plant Molecular Biology Reporter 17(1): 74-74; doi:10.1023/A:1007528101452
Kanupriya, Shobhana M, Vasugi C, Aswath C, Radhika V, Reddy L, Dinesh M (2012) Genetic relationship among papaya (Carica papaya) and wild papaya (Vasconcellea species) using RAPD and ISSR marekers. Indian Journal of Agricultural Sciences 82(4): 366-369
Kyndt T, Haegeman A, Glabeke SV, Maertens I, Droogenbroeck BV, Roldán-Ruiz I, Gheysen G (2005) Isolation and characterization of microsatellite loci in the highland papaya Vasconcellea x heilbornii V Badillo (Caricaceae). Molecular Ecology Notes 5(3): 590-592; doi:10.1111/j.1471-8286.2005.00999x
Leva A, Petruccelli R (2012) Monitoring of cultivar identity in micropropagated olive plants using RAPD and ISSR markers. Biol Plant 56(2): 373-376; doi:10.1007/s10535-012-0102-6
Martins M, Sarmento D, Oliveira M (2004) Genetic stability of micropropagated almond plantlets as assessed by RAPD and ISSR markers. Plant Cell Rep 23(7): 492-496; doi:10.1007/s00299-004-0870-3
Mohanty S, Panda M, Sahoo S, Nayak S (2011) Micropropagation of Zingiber rubens and assessment of genetic stability through RADP and ISSR markers. Biol Plant 55(1): 16-20; doi:10.1007/s10535-011-0002-1
Mogdil M, Mahajan K, Chakrabarti S, Sharma D, Sobti R (2005) Molecular analysis of genetic stability in micropropagated Apple rootstock MM106. Sci Hortic 104(2): 151-160; doi:10.1016/j.scienta.2004.07.009
Nayak S, Kaur T, Mohanty S, Ghosh G, Choudhury R, Acharya L, Subudhi E (2011) In vitro and ex vitro evaluation of long-term micropropagated turmeric as analyzed through cytophotometry, phytoconstituents, biochemical and molecular markers. Plant Growth Reg 64(1): 91-98; doi:10.1007/s10725-010-9541-2
Ocampo JP, Dambier D, Ollitrault P, Coppens G, Brottiers P, Froelicher Y, Risterucci A (2006) Microsatellite markers in Carica papaya L.: isolation, characterization and transferability to Vasconcellea species. Molecular Ecology Notes 6(1): 212-217; doi:10.1111/j.1471-8286.2006.01197.x
Ochoa V, Jiménez P, Proaño K, Segovia-Salcedo C (2008) Genética Poblacional de Polylepis pauta y P. incana en el Páramo de Papallacta. Revista ESPE Ciencia y Tecnología 1(1): 71-79
Palombi M, Damiano C (2002) Comparison between RAPD and SSR molecular markers in detecting genetic variation in kiwifruit (Actinidia deliciosa A. Chev). Plant Cell Rep 20(11): 1061-1066; doi:10.1007/s00299-001-0430-z
Parida R, Mohanty S, Nayak S (2011) Evaluation of genetic fidelity of in vitro propagated greater galangal (Alpinia galanga) using DNA based markers. Int J Plant Animal Environ Sci 1(3): 123-133
Peakall R, Smouse PE (2006) GENALEX 6: genetic analysis in Excel Population genetic software for teaching and research. Molecular Ecology Notes 6(1): 288-295; doi:10.1111/j.1471-8286.2005.01155.x
Ramakrishnan M, Ceasar S, Duraipandiyan V, Ignacimuthu S (2014) Efficient plant regeneration from shoot apex explants of maize (Zea mays) and analysis of genetic fidelity of regenerated plants by ISSR markers. Plant Cell Tiss Organ Cult 119(1): 183-196; doi:10.1007/s11240-014-0525-1
Ramírez M, Iglesias L (2015) Indirect organogenesis and assessment of somaclonal variation in plantlets of Vanilla planifolia Jacks. Plant Cell Tiss Organ Cult 123(3): 657-664; doi:10.1007/s11240-015-0868-2
Rathore NS, Rai MK, Phulwaria M, Rathore P, Shekhawat NS (2014) Genetic stability in micropropagated Cleome gynandra revealed by SCoT analysis. Acta Physiol Plant 36(2): 555-559; doi:10.1007/s11738-013-1429-0
Robles A, Salinas D, Armijos W, Sánchez A, Torres R (2013) Estudio de la Variabilidad Morfológica de Aislados Fúngicos Asociados con la Enfermedad de la Marchitez Vascular del Babaco (Vasconcellea x heilbornii var. pentagona). Centro de Biotecnología 2(1): 34-44
Rout G, Senapati S, Aparajita S, Palai S (2009) Studies on genetic identification and genetic fidelity of cultivated banana using ISSR markers. Plant Omics J 2(6): 250-258
Saker M, Adawy SS, Mohamed A, El-Itriby HA (2006) Monitoring of cultivar identity in tissue culture-derived date palms using RAPD and AFLP analysis. Biol Plant 50(2): 198-204; doi:10.1007/s10535-006-0007-3
Sharma S, Pamidimarri D, Anand K, Reddy M (2011) Assessment of genetic stability in micropropagules of Jatropha curcas genotypes by RAPD and AFLP analysis. Ind Crop Prod 34(1): 1003-1009; doi:10.1016/j.indcrop.2011.03.008
Schelderman X, Kyndt T, d´Eeckenbrugge G, Ming R, Drew R, Van Droogenbroeck B, Van Damme P, Moore P (2011) Vasconcellea. En: Kole C (Ed). Wild crop relatives: genomic and breeding resources, tropical and subtropical fruits, pp. 213-249. Springer-Verlag Berlin Heidelberg, Berlin; ISBN: 978-3-642-20446-3
Vega R, Kitto S (1991) Regeneration of Babaco (Carica pentagona) from Ovular callus. United States. Journal of the American Society for Horticultural Science 116(4): 747-752
Vélez-Mora DP, Aemijos GR, Jordán MZ (2015) Enhancement of germination, hyperhydricity control and in vitro shoot formation of Vasconcellea stipulata Badillo. Revista Colombiana de Biotecnología XVIII(2): 16-21; doi:10.15446/rev.colomb.biote.v17n2.43611
Yin ZF, Zhao B, Bi WL, Chen L, Wang QC (2013) Direct shoot regeneration from basal leaf and of Lilium and assessment of genetic stability in regenerates by ISSR and AFLP markers. In vitro Cell Dev Biol Plant 49(2): 333-342; doi:10.1007/s11627-013-9501-4
Recibido: 22-03-2017
Aceptado: 31-05-2017
Copyright (c) 2017 Biotecnología Vegetal
Biotecnología Vegetal eISSN 2074-8647, RNPS: 2154. ISSN 1609-1841, RNPS: 0397 Editada por: Instituto de Biotecnología de las Plantas. Universidad Central Marta Abreu de Las Villas. Carretera a Camajuaní km 5.5, Santa Clara, Villa Clara, Cuba CP 54 830 Tel: 53 42200124, e-mail: info@ibp.co.cu

Biotecnología Vegetal está bajo una Licencia Creative Commons Atribución-NoComercial 4.0 Internacional.
