Reseña bibliográfica
Biotecnología Vegetal Vol. 18, No. 2: 63 - 80, abril - junio, 2018
Instituto de Biotecnología de las Plantas. UCLV. MES.
eISSN 2074-8647, RNPS: 2154
Estrategias para la selección in vitro de plantas transgénicas de Digitalis L.
Approaches for in vitro selection of genetically modified Digitalis L. plants
Elizabeth Kairuz1,2, Naivy Pérez-Alonso2,3, Borys Chong-Pérez2,4
1Departamento de Biología, Facultad de Ciencias Agropecuarias, Universidad Central Marta Abreu de Las Villas. Carretera a Camajuaní km 5,5. Santa Clara. Villa Clara. Cuba. CP 54 830. e-mail: kairuzhd@uclv.edu.cu
2Instituto de Biotecnología de las Plantas, Universidad Central Marta Abreu de Las Villas. Carretera a Camajuaní km 5,5. Santa Clara. Villa Clara. Cuba. CP 54 830.
3Botanical Solutions SpA. Ave Quilin 3550. Macul. Santiago de Chile. Chile. CP 7810000.
4Sociedad de Investigación y Servicios BioTECNOS Ltda. Camino a Pangal km 2,5. San Javier. Linares. Chile. CP 3660000.
Resumen
Las plantas del género Digitalis son reconocidas por sus propiedades medicinales debido a su capacidad para producir cardenólidos, como la digoxina. Estos compuestos son glucósidos cardiotónicos insustituibles para el tratamiento de enfermedades cardiovasculares. Además, ha adquirido valor agregado debido a su aplicación potencial en el tratamiento de varios tipos de cáncer. La ingeniería metabólica es una prometedora herramienta biotecnológica para incrementar la biosíntesis de cardenólidos in vitro. Dentro del proceso de transformación genética la diferenciación de las plantas transformadas, es un paso clave para alcanzar su eficiencia. El objetivo de esta revisión fue sintetizar los principales aspectos a tener en cuenta para realizar un esquema de selección eficiente en este género. Incluye un compendio de las características de los genes marcadores de selección más utilizados, los principios para la selección de eventos transgénicos, un análisis de los agentes selectivos empleados en el género y su eficacia en la transformación genética. Finalmente, se exponen las perspectivas de investigación en el tema.
Palabras clave: antibiótico, cardenólidos, genes marcadores de selección, hpt, npt II
Abstract
Medicinal plants of Digitalis genus are well known for its ability to produce cardenolides as digoxin. These cardiotonic glucosides are irreplaceable for heart failure treatment. In addition, cardenolides have acquired added value due to its potential application on several types of cancer treatment. Metabolic engineering is a promising biotechnological tool to enhance cardenolides biosynthesis in vitro. The differentiation of transformed plants is a key step to achieve efficiency in genetic transformation process. The aim of this review was to synthesize the main aspects to make an efficient selection scheme in Digitalis L. It includes a compendium of most used selection marker genes features, main principles for transgenic events selection, an analysis of selective agents used in the genus and their effectiveness in genetic transformation. Finally, perspectives of research in the subject are exposed.
Keywords: antibiotics, cardenolides, hpt, nptII, selection marker genes
Introducción
El desarrollo de la industria farmacéutica y las ciencias de la salud, posibilitan el incremento de la calidad de vida en la población mundial (Mathers et al., 2015; Suzman et al., 2015). En este sentido, existe una búsqueda continua de compuestos de origen natural o de alternativas para su producción, con el objetivo de emplearlos en el tratamiento de enfermedades de alta incidencia (David et al., 2015).
Desde 1950 las enfermedades cardiovasculares han sido la primera causa de muerte en humanos, seguidas del cáncer. Aunque en la última década se ha identificado una tendencia a la disminución de las primeras y un aumento considerable de las muertes por neoplasias, hasta igualarlas o superarlas en algunas poblaciones (Heron y Anderson, 2016).
Los cardenólidos, como la digoxina, son glucósidos cardiotónicos imprescindibles para el tratamiento de las insuficiencias cardiacas y arritmias supraventriculares, que disminuyen el ingreso en hospitales de pacientes que los consumen (Ziff et al., 2015). En la última década se ha potenciado el estudio del efecto antiproliferativo y apoptótico de los cardenólidos sobre las células cancerígenas. Los resultados positivos en la regulación de la proliferación celular de tejidos normales y varios tipos de neoplasias, han llevado al desarrollo de ensayos clínicos con digoxina en pacientes con cáncer (Verma et al., 2016).
En consecuencia, existe una alta demanda internacional de estos compuestos bioactivos, que deben ser importados en los países que no pueden cultivar estas plantas, dentro de los cuales se incluye Cuba. Estos metabolitos secundarios se producen por plantas del género Digitalis L., las cuales constituyen la única fuente viable para su obtención a escala industrial (Sales et al., 2011).
El alto valor económico de las plantas de Digitalis, debido a la explotación por la industria farmacéutica, provoca pérdidas en las poblaciones silvestres, ya sea por su cultivo extensivo o el desarrollo de acciones insuficientes para recuperarlas. Los ciclos bienales de la planta y las limitaciones bióticas y abióticas en su cultivo, hacen que sus semillas tengan una baja frecuencia de germinación, y se limite su propagación. Además, la cosecha incontrolada del material vegetal de especies de Digitalis antes del proceso de floración, provoca que las poblaciones desaparezcan rápidamente (Verma et al., 2016).
Por otro lado, tanto la producción como el contenido de cardenólidos pueden variar en respuesta a las diferentes estaciones del año y condiciones ambientales (Roca-Pérez et al., 2004).
Con el objetivo de minimizar estas limitaciones, se han desarrollado varias estrategias biotecnológicas para propagar plantas de alta calidad, con una producción uniforme en condiciones controladas. El cultivo in vitro de especies de Digitalis ha sido extensamente investigado en este sentido, y ha permitido dilucidar que la biosíntesis de los cardenólidos se produce sólo en tejidos verdes diferenciados (Stuhlemmer y Kreis, 1996).
Basados en estos conocimientos, se obtuvieron niveles apreciables de compuestos digitálicos, mediante la propagación de plantas de Digitalis purpurea L. en sistemas de inmersión temporal, aunque todavía menores que los producidos en condiciones de campo (Pérez-Alonso et al., 2009). También se ha desarrollado la elicitación (Pérez-Alonso et al., 2012, Pérez-Alonso et al., 2014a) y protocolos de transformación genética vía Agrobacterium tumefaciens L., de segmentos foliares de plantas cultivadas in vitro (Li et al., 2014; Pérez-Alonso et al., 2014b). Estos últimos podrían posibilitar la expresión de genes involucrados en la síntesis de cardenólidos, que potencien su producción. La aplicación de varias de estas estrategias, permitirá propagar masivamente plantas transformadas con una productividad más elevada y uniforme que la registrada en condiciones naturales, que supla las necesidades de cardenólidos de cada país. Un paso clave para desarrollar un protocolo de transformación genética eficiente, es el establecimiento de las condiciones de selección del tejido transformado.
Debido a la importancia del cultivo in vitro de Digitalis, en menos de una década se han realizado varias revisiones que abordan aspectos importantes como los métodos de cultivo, el metabolismo de los cardenólidos, la seguridad y eficacia del empleo de digoxina, sus aplicaciones medicinales, entre otros aspectos (Sales et al., 2011; Kreis y Muller-Uri, 2013; David et al., 2015; Martín et al., 2015; Verma et al., 2016). Sin embargo, en ninguno de estos artículos se consideran los resultados en la selección in vitro de plantas transformadas del género Digitalis. Por ello, el objetivo de esta revisión fue sintetizar los principales aspectos a tener en cuenta para realizar un esquema de selección eficiente en este género. Para ello se compendian las características de los genes marcadores de selección más utilizados, los principios para la selección de eventos transgénicos, los agentes selectivos empleados en el género y su eficacia. Finalmente, se exponen las perspectivas para la selección y obtención de plantas transgénicas de Digitalis que permitan incrementar la producción de cardenólidos in vitro.
GENES MARCADORES DE SELECCIÓN
El desarrollo de un protocolo de transformación genética requiere de un método de selección de las plantas transformadas. Esta diferenciación disminuye considerablemente la laboriosidad y el consumo de recursos asociados a procesar todo el material vegetal transformado, hasta que se realice su análisis molecular. Por tanto, es muy común la utilización de genes marcadores de selección (GMS) en los vectores de transformación genética. Estos pueden conferir resistencia a herbicidas, antibióticos, u otras condiciones de toxicidad o estrés para la planta (Rosellini, 2011), lo que permite diferenciar aquellas células, tejidos y plantas que han sido transformados de los que no se modificaron genéticamente.
Los GMS pueden clasificarse en dependencia de si permiten el desarrollo del tejido transformado (selección positiva) o causan la muerte de este (negativa). También pueden ser clasificados como condicionales o no, en dependencia de si la selección requiere la presencia de sustratos externos o no (Breyer et al., 2014).
Los marcadores de selección más comúnmente utilizados son positivos y condicionales, como los genes de resistencia a antibióticos. Varias ventajas hacen factibles el empleo de estos como agentes selectivos entre las más notables se destaca que son efectivos, tienen una elevada eficiencia ganancia-costo y son aplicables en numerosas especies (Breyer et al., 2014). Sin embargo, la introducción de transgenes de resistencia a antibióticos ha generado preocupaciones éticas asociadas a los riesgos de que ocurra transferencia génica horizontal, sobre todo si se emplean también con fines clínicos.
Los GMS más utilizados fueron aislados de las bacterias Klebsiella pneumoniae (Schroeter) Trevisan y Escherichia coli (Migula) Castellani y Chalmers. El gen que codifica para la neomicina fosfotransferasa (nptII) confiere resistencia a los antibióticos aminoglucósidos tales como kanamicina, neomicina, paromomicina y geneticina (G418) (Bevan et al., 1983; Fraley et al., 1983; Herrera-Estrella et al., 1983). Por otra parte, el gen de la higromicina fosfotransferasa (hpt) confiere resistencia a higromicina B, un aminoglucósido tóxico a la célula vegetal debido a que inhibe la síntesis proteica (Blochlinger y Diggelmann, 1984).
El gen nptII fue el primero que se empleó como marcador de selección en plantas. Desde entonces es el sistema de selección más comúnmente aplicado para generar plantas transgénicas con propósitos científicos (Miki y McHugh, 2004; Breyer et al., 2014).
Diversos estudios se han desarrollado con el objetivo de dilucidar los efectos predecibles e inesperados del empleo de GMS, mas, en el caso del nptII los resultados son favorables. El producto de este gen no es una toxina o alergeno para animales o humanos, ni su uso en plantas transgénicas compromete el empleo de antibióticos en estos organismos. Adicionalmente, es poco probable que se transfiera del genoma de las plantas a otros microorganismos por transferencia génica horizontal, es altamente sensible a hidrólisis enzimática y no genera efectos pleiotrópicos (Ramessar et al., 2007; Miki et al., 2009). Por estas razones los cultivos transgénicos en los cuales se ha introducido este gen, han sido aprobados para ensayos de campo y la comercialización (Breyer et al., 2014). Por ejemplo, en el año 2011 fue empleado para la selección de cultivos transgénicos en más de 1100 publicaciones científicas de la Web de Ciencias (Rosellini, 2012) y esta tendencia continúa en la actualidad (Hanana et al., 2018).
Higromicina B es el segundo antibiótico de uso más frecuente en el proceso de selección de eventos transgénicos, debido en parte a su alta toxicidad para las plantas. Es usual su empleo como GMS cuando el gen nptII no ha sido efectivo (Rosellini, 2012). En análisis de una muestra de publicaciones durante el año 2002, este gen fue utilizado en hasta un 30% de las plantas transgénicas obtenidas con fines investigativos (Miki y McHugh, 2004). Su aplicación en cotiledones de remolacha azucarera (Beta vulgaris L.) generó un efecto tóxico entre 20 y 30 veces superior a kanamicina y neomicina, respectivamente (Joersbo y Okkels, 1996). También ha sido mencionado en estudios más recientes, como el agente selectivo más efectivo en la selección de callos de Oriza sativa L. (Wakasa et al., 2007) y Elaeis guineensis Jacq. (Parveez y Majid, 2018).
Otro ejemplo de selección positiva condicional es el uso de herbicidas como agentes selectivos. Estos se emplean con fines agronómicos o en cultivos naturalmente resistentes a antibióticos. Este sistema tiene la ventaja de que las plantas pueden ser asperjadas para la selección en casa de cultivo o en campo y a su vez puede ser el gen de interés. Sin embargo, posee desventajas entre las que se incluyen su toxicidad y alergenicidad, que pueden causar daños a la salud humana y a la seguridad de nuevos productos comestibles, así como potenciales efectos negativos en organismos que no son su diana (Ramessar et al., 2007). También pueden generar resistencia a herbicidas en otros cultivares o especies compatibles sexualmente, lo que limita las opciones de control para los agricultores y el uso de herbicidas con fines no científicos. La mayoría de las plantas genéticamente modificadas que han sido probadas en campo o aprobadas para la comercialización, contienen GMS de resistencia a herbicidas. Entre los más utilizados se encuentra el gen bar de Streptomyces hygroscopicus (Jensen) Waksman y Henrici, que le permite resistir el efecto de la fosfinotricina. Este compuesto constituye el ingrediente activo de numerosos herbicidas comerciales como el BastaTM (Breyer et al., 2014).
Los genes de selección negativa generalmente se utilizan en combinación con genes de selección positiva, cuando se desea escindir los GMS o disminuir el quimerismo (Rosellini, 2012). Para su aplicación se utilizan construcciones genéticas que contengan entre los sitios de escisión, un gen que posibilite la selección positiva (como los genes de resistencia a antibiótico) y otro de selección negativa. Por lo tanto, se escogen las células transformadas resistentes y cuando se active la escisión se transfieren los explantes al medio de cultivo con el compuesto tóxico para las células transformadas. Si la escisión es exitosa se excluyen del genoma ambos GMS y sobrevive el tejido. De lo contrario, se eliminan los explantes que mantienen los transgenes selectivos (Breyer et al., 2014).
Los genes de selección negativa provienen de bacterias o virus, por lo que su toxicidad puede variar en diferentes cultivos. Aunque han sido empleados exitosamente para la selección negativa en plantas, siempre es necesario determinar su efecto cuando se va a usar con nuevas especies (Majhi et al., 2014).
De igual modo, los genes de selección negativa pueden ser condicionales o no. Cuando no se requiere de un sustrato externo, las células transgénicas se someten inmediatamente al efecto letal del agente selectivo. Este es el caso de genes codificantes para el fragmento A de la toxina de Corynebacterium diphtheriae (Kruse) Lehmann y Neumann (DT-A) o la endotoxina A de Pseudomonas aeruginosa (Schroeter) Migula (Osakabe et al., 2014).
El gen codA de E. coli codifica para la enzima citosina desaminasa (EC 3.5.4.1), esta cataliza la conversión de 5-fluorocitosina (5-FC) a 5-fluorouracilo, el cual es una sustancia citotóxica debido a que inhibe la síntesis de ácidos nucleicos (de Oliveira et al., 2015). A pesar de ser el gen más empleado para el desarrollo de estrategias de selección negativa condicional, no es posible su utilización en todos los cultivos, pues en algunos casos no se produce toxicidad o se requiere de concentraciones muy elevadas de 5-FC para que sea efectivo (Chong-Pérez et al., 2012; Osakabe et al., 2014).
Los genes reporteros también se incluyen usualmente en los vectores de transformación genética, pues los productos de su expresión pueden ser visualizados. Esta cualidad es aplicada para establecer y optimizar protocolos de transformación genética, comparar los patrones de expresión de los promotores introducidos y reconocer el tejido transformado (Deliolanis et al., 2014; Nietsch et al., 2017; Lamont et al., 2017). También son útiles cuando hay muchos escapes del proceso de selección. Estos se clasifican en vital o no, en dependencia de si se pueden observar en el tejido sin afectar su viabilidad o no, así como en cuanto a la necesidad de un sustrato o sustancia adicional para su detección (condicional o no condicional). La selección del gen reportero está determinada por su estabilidad, período de actividad y las señales de fondo que pueden interferir en su observación (Kavita y Burma, 2008).
Los genes reporteros se han empleado como marcadores visuales de la expresión génica y la localización de proteínas in vivo, en un amplio espectro de organismos tanto procariontes como eucariontes. Sin embargo, la detección de la expresión de los productos génicos como la ß-galactosidasa (lacZ) (Helmer et al., 1984), ß-glucuronidasa (Jefferson et al., 1987) y luciferasa (luc) (Gallie et al., 1989), requiere de sustratos exógenos/co-factores, lo que conlleva a que su aplicación se vea limitada (Sheen et al., 1995).
El gen uidA o gusA codifica para la enzima β-glucuronidasa (EC 3.2.1.128) que produce un complejo de inclusión azul al reaccionar con sus sustratos (actividad GUS). Es uno de los genes reporteros más empleados y está presente en varias plantas modificadas que han sido aprobadas para su comercialización (Breyer et al., 2014). Sin embargo, la visualización de la actividad GUS está limitada por la difusión del sustrato a los tejidos donde se produce la enzima. Su cuantificación mediante ensayos fluorométricos no destructivos es laboriosa y poco factible cuando se deben analizar grandes cantidades de material vegetal transformado. Por esta razón, comúnmente se procede a incubar el explante transformado con el sustrato y posteriormente se elimina la clorofila u otros pigmentos que interfieran con la visualización de este producto azul en el tejido. Esta práctica destruye la vitalidad del tejido y posibilita evaluar la actividad GUS en el explante solo una vez. A pesar de estas desventajas, es el gen reportero más popular en los informes de transformación genética en plantas (Rosellini, 2012).
Un gen reportero inerte que posea una señal intrínseca tal como una proteína bioluminiscente, puede ser aplicado como marcador celular universal (Prasher et al., 1992). Por ejemplo, la proteína verde fluorescente (GFP) fluoresce con un máximo de excitación a 395 nm y un mínimo a 475 nm, y emite una señal detectable a 509 nm en el espectro visible. Para su emisión requiere luz ultravioleta y oxígeno, pero no es necesario el empleo de un sustrato exógeno. Esta propiedad le confiere una gran aplicabilidad en tejidos vivos, ya que se expresa de manera autónoma e independiente del tipo de célula y su localización.
Diferentes mutaciones en la secuencia aminoacídica de la proteína verde fluorescente han originado nuevas variantes con transformaciones en el espectro de excitación y emisión, así como niveles de emisión superiores que conducen a un aumento de la fluorescencia al incidir luz ultravioleta sobre esta (Shaner et al., 2005). Entre las variantes más empleadas se encuentra la proteína verde fluorescente potenciada (egfp), con propiedades superiores asociadas con el aumento de la fluorescencia y la fotoestabilidad. La principal limitación del empleo de proteínas fluorescentes, es la necesidad de un equipamiento específico y costoso para su detección.
Una alternativa prometedora, es el empleo de genes de factores de transcripción involucrados en la síntesis de antocianinas. Estos permiten que las plantas genéticamente modificadas sean identificadas a simple vista, no requieren la adición de sustratos, no son tóxicas, ni afectan el desarrollo. Adicionalmente, el consumo de antocianinas es beneficioso para la salud y se espera que no existan dificultades para su comercialización (Breyer et al., 2014).
Otras características de los GMS y alternativas para su empleo han sido ampliamente revisadas en publicaciones anteriores (Miki y McHugh, 2004; Ramessar et al., 2007; Rosellini, 2012; Breyer et al., 2014).
De forma general, la selección de estos genes depende de diversos factores entre los que se incluyen las particularidades del cultivo, el esquema de selección y el protocolo de transformación genética previamente propuesto. Además, influye la construcción genética utilizada y el objetivo de la modificación genética.
PRINCIPIOS PARA ESTABLECER UN ESQUEMA DE SELECCIÓN EFICIENTE
La eficiencia de selección (SE) puede ser definida como el número de eventos de selección independientes en 100 eventos de regeneración, siendo 100-SE la tasa de escapes (Rosellini, 2012). Por tanto, el GMS empleado debe posibilitar ejercer la presión necesaria para detener el crecimiento de las células no transformadas, y permitir solo la proliferación del tejido preferido, con una constitución genética homogénea.
Existen numerosos aspectos a tener en cuenta para establecer un esquema de selección eficiente, los cuales se prefijan en primer lugar por el protocolo de regeneración propuesto. Este delimita la composición del medio de cultivo, la frecuencia de subcultivo, las condiciones abióticas y los tipos de explante. Partiendo de estas características, se debe escoger el GMS, el agente selectivo, el momento y la duración de la selección.
La composición del medio de cultivo, la frecuencia de subcultivo y las condiciones abióticas pueden influir sobre el proceso de selección, principalmente cuando se requieren de sustratos externos, como antibióticos, para que ocurra la diferenciación del tejido. Para ello se debe tener en cuenta la estabilidad de estos aditivos y la capacidad de reaccionar con componentes del medio de cultivo.
La estructura, función y efectos de los antibióticos aminoglucosídicos en plantas cultivadas in vitro y protocolos de transformación genética, ha sido revisado por Padilla y Burgos (2010). De acuerdo con lo planteado por estos autores, para elaborar un sistema de selección efectivo con este tipo de compuestos es necesario analizar: i) el modo de acción del agente selectivo en células vegetales cultivadas in vitro, ii) la complejidad del explante sometido a selección y iii) las características del protocolo de transformación genética.
La susceptibilidad de las plantas a antibióticos de naturaleza aminoglucosídica es variable para las diversas especies, cultivares y tejidos. Las diferencias en la sensibilidad a un mismo antibiótico en cotiledones, hipocótilos, discos foliares y callos, son perceptibles si se tiene en cuenta las diferencias en su naturaleza química. Mientras mayor y más compleja sea la estructura de un explante, más difícil será determinar la fitotoxicidad de estos compuestos, ya que existirán mecanismos que prevengan su llegada a la célula. Teniendo en cuenta estos aspectos, es aconsejable aplicar el agente selectivo inmediatamente después del proceso de transformación y antes de la regeneración de las plantas. Lo primero impide la proliferación de células no transformadas antes de la multiplicación del tejido, pues en las plantas las concentraciones necesarias para alcanzar dosis inhibitorias son más altas debido a la complejidad del explante. La eficiencia de selección con un determinado agente, puede variar de acuerdo con el protocolo de regeneración seleccionado. El origen pluricelular y la complejidad estructural de los brotes, hace que sea más probable obtener escapes en protocolos de regeneración vía organogénesis directa, que cuando se produce la formación de callos (Padilla y Burgos, 2010).
Consecuentemente, es necesario establecer estos parámetros para cada medio de cultivo usado en el protocolo; lo cual evitaría los comunes escapes y formaciones de quimeras referenciados en múltiples trabajos de transformación. También deben realizarse al menos dos subcultivos de los explantes en el medio de cultivo selectivo. Este puede efectuarse cada aproximadamente 15 días, para mantener la concentración del antibiótico y extender su efecto sobre el tejido o de acuerdo con los periodos de subcultivo propuestos en el protocolo de propagación, siempre que se conserve el efecto del agente selectivo. En ocasiones no es posible observar los efectos inhibitorios, si no se prolonga la duración de la presión de selección (Kairuz et al., 2013).
De cualquier modo, se ha sugerido que el agotamiento del antibiótico en la vecindad de las células transgénicas, permite la proliferación de células no transformadas y la regeneración de brotes. En consecuencia, la mayoría de los sistemas de transformación genética, es predecible la obtención de un número de escapes y quimeras, a pesar del correcto diseño del proceso de selección (Padilla y Burgos, 2010).
Finalmente, para escoger un GMS no solo se debe tener en cuenta su efectividad porque resulte en un buen esquema de selección. La principal propiedad de un GMS condicional positivo no es que permita sobrevivir células altamente selectivas con resistencia a un sustrato, sino que potencie el crecimiento selectivo y la diferenciación de estos tejidos (Breyer et al., 2014). Esta condición no se suele evaluar cuando se comparan los GMS o los agentes selectivos. Atendiendo a ello, es recomendable tener en cuenta la velocidad de crecimiento o el coeficiente de multiplicación del tejido seleccionado, para el correcto establecimiento de un esquema de selección de este tipo. Este aspecto también es válido para el empleo de otros tipos de GMS.
TRANSFORMACIÓN GENÉTICA Y SELECCIÓN IN VITRO DE PLANTAS DE DIGITALIS L.
En la mayoría de las publicaciones en las que se transfieren GMS o se proponen esquemas de selección en el género Digitalis, se utilizan los genes nptII y uidA. Sin embargo, hay diferencias entre ellas, en cuanto a la vía de transformación genética, el tipo de explante sometido a selección o el agente selectivo, entre otros aspectos.
En los primeros trabajos de transformación genética realizados se emplearon cepas de Agrobacterium rhyzogenes (Riker) Conn. Mas, la imposibilidad o la baja eficiencia de regeneración de las raíces pilosas, condujeron a los investigadores a realizar la transferencia con otra especie del género: Agrobacterium tumefaciens. Para ello, se emplearon cepas silvestres, que luego fueron sustituidas por cepas modificadas con vectores auxiliares y genes de interés.
El tipo de explante más empleado son segmentos foliares, aunque también se ha explorado brevemente la transformación de cloroplastos, ápices y colonias celulares embriogénicas. En muchos casos se realiza la regeneración vía organogénesis directa o indirecta. La primera es más rápida, pero puede implicar mayor aparición de escapes y quimeras (Sales et al., 2003).
Entre los agentes selectivos, kanamicina y geneticina han sido los más usuales, aunque también han surgido antecedentes para la selección con fosfinotricina e higromicina B. Por lo general, durante el proceso de selección se realizan al menos dos subcultivos cada 15 días (Tabla 1), lo que no imposibilita la aparición de escapes, principalmente si se emplea kanamicina y un protocolo de regeneración vía organogénesis directa (Sales et al., 2002; Sales et al., 2003).
Hasta el presente, el gen uidA se incluye en todos los estudios de transformación genética de Digitalis (Tabla 1), como una vía para confirmar su éxito, a partir de la comprobación de la actividad GUS. No se han explorado otros genes reporteros.
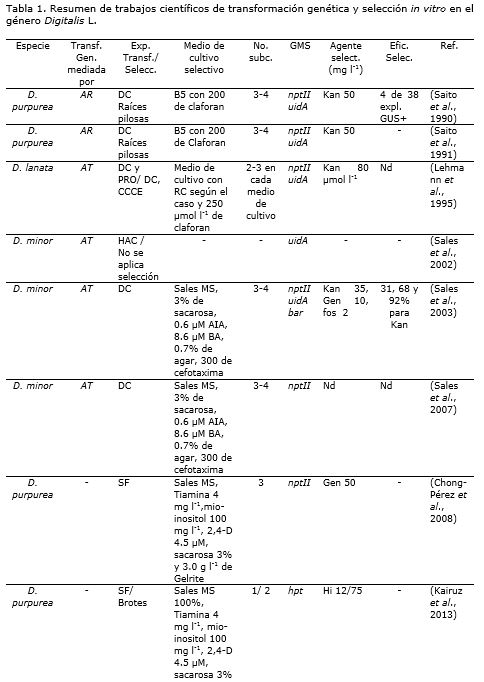
A continuación se analizan en orden cronológico, los principales trabajos de transformación genética y selección en el género Digitalis. Estos y otros aspectos se resumen en la tabla 1.
Transformación genética vía Agrobacterium rhyzogenes
Los estudios de transformación genética y selección en el género Digitalis se comenzaron a publicar en la década del 1990. Inicialmente la transferencia de genes fue mediada por Agrobacterium rhizogenes, con el empleo de dos vectores binarios para la inducción de raíces (Saito et al., 1990). En este trabajo se introducen en el genoma de D. purpurea los transgenes nptII y uidA. Mas, los autores no consideraron necesario realizar la selección de los discos foliares transformados, dada la alta frecuencia de transformación del protocolo propuesto. Al obtenerse las raíces pilosas, estas eran transferidas a medio de cultivo B5 (Gamborg et al., 1968) con 200 mg l-1 de cefatoxima.
Posteriormente, los explantes a los que se transfirió el ADN-T del plásmido pGSGluc1, luego de 2-3 semanas, formaron raíces pilosas delgadas, que fueron seleccionadas con kanamicina 50 mg l-1 en medio de cultivo B5. De los 38 clones de raíces escindidos y transferidos al medio de cultivo B5, diez fueron capaces de crecer en kanamicina. De estos, en seis se detectó la presencia de agropinas y manopinas, de los cuales cuatro resultaron positivos al ensayo enzimático con β-glucuronidasa (GUS). En los experimentos donde se empleó el plásmido pBI121, se procedió de forma similar, pero sin adicionar kanamicina al medio de cultivo B5. Se analizaron 27 clones de raíces, de los cuales en ocho se detectaron opinas y en tres el ensayo enzimático GUS fue positivo. Cinco de los siete clones positivos a ambos ensayos, fueron escogidos por su rápido crecimiento, para continuar el análisis de la integración y expresión de los transgenes. Todos ellos mostraron la presencia del gen uidA en el genoma por Hibridación de Southern y los productos de la actividad enzimática GUS y neomicina fosfotransferasa II (NPT-II), a partir de la extracción de proteínas totales. También se realizó el ensayo histoquímico GUS, que resultó en la tinción de los tejidos vasculares de todos los transformantes analizados. Por último, en las raíces verdes transformadas, se determinó la presencia de cardenólidos por ELISA (Saito et al., 1990).
En este primer acercamiento a la ingeniería genética de Digitalis para potenciar la producción de metabolitos secundarios de interés farmacológico, Saito et al. (1990) destacaron varias ventajas. Estas implican la posibilidad de obtener raíces transgénicas sin el empleo de agentes selectivos, la factibilidad de emplear este protocolo para potenciar la producción de cardenólidos mediante la manipulación genética y el hecho de que algunas especies de plantas pueden regenerarse a partir de las raíces. Previamente, estos autores realizaron estudios preliminares para la regeneración de brotes, con diferentes combinaciones de reguladores de crecimientos, pero sin éxito (Saito et al., 1990). En la discusión de estos resultados, no se valora la eficiencia de selección o su importancia para la transformación genética a mayor escala. Tampoco el hecho de que incluso cuando se utilizan 50 mg l-1 de kanamicina, se producen escapes de este proceso.
La continuidad de este trabajo pionero en la transformación genética de D. purpurea, fue publicada un año después, e incluyó otras dos especies de interés medicinal: Nicotiana tabacum L. y Glycyrrhiza uralensis Fischer (Saito et al., 1991). En este caso el objetivo de la investigación fue determinar el modo de expresión génica del promotor dual TR 1´-2´ sobre los genes nptII y uidA, respectivamente. Para ello, estudiaron la especificidad de tejido y las señales de estrés fisiológico sobre la expresión del promotor TR, en estas tres especies de plantas transgénicas. Como señales de estrés se provocaron lesiones en los explantes, o se evaluaron los efectos de su inmersión en cada uno de los seis reguladores de crecimiento vegetal, a una concentración de 10 mg l-1.
En el caso particular de D. purpurea, emplearon una de las líneas transgénicas seleccionadas y analizadas en el trabajo anterior. También las mismas técnicas para confirmar la integración y expresión de los genes nptII, uidA y la presencia de opinas. Por ello no contribuyó con un nuevo aporte a los conocimientos sobre la metodología de transformación y selección de esta especie, pero sí sobre la expresión de los transgenes en las raíces pilosas. Mediante la tinción histoquímica de un corte transversal de las raíces pilosas de D. purpurea, fue posible observar la expresión fuerte del gen uidA en el floema y los tejidos circundantes. También se demostró que las lesiones en los explantes potencian considerablemente la expresión de este gen bajo el control del promotor TR 2´, en contraste con el efecto de los reguladores de crecimiento, con los cuales no se observaron diferencias (Saito et al., 1991).
Por último, Saito et al. (1991) analizaron la importancia del uso de un promotor fuerte que permitiera la expresión sitio-específica de transgenes en el floema de la planta, para modificar rutas metabólicas secundarias. Teniendo en cuenta que la mayoría de los metabolitos secundarios se transportan del tejido donde se sintetizan, hacia órganos especializados en el almacenamiento o la transformación metabólica (Saito et al., 1991). En consecuencia, la obtención de plantas transgénicas con un promotor de expresión fuerte en floema, podría permitir la biotransformación específica de metabolitos secundarios de interés.
Años después se publicaron nuevos resultados en la transformación genética de este género vía A. rhyzogenes, pero en este caso en Digitalis lanata Ehrh (Pradel et al., 1997). Este constituye el primer informe de regeneración de brotes y plantas a partir de raíces pilosas de esta especie, aunque con una alta hiperhidricidad y aparición de plantas con bajo contenido de clorofila y hojas deformadas. En este trabajo se utilizaron cepas silvestres de A. rhyzogenes, por lo que no se introdujeron GMS.
Debido a la baja eficiencia de regeneración de plantas a partir de raíces pilosas y el hecho de que los cardenólidos se producen asociados a tejidos verdes (Sales et al., 2011), los intentos de transformar plantas de Digitalis vía A. rhyzogenes fueron sustituidos por los prometedores resultados de otras investigaciones en las cuales se utiliza A. tumefaciens.
Transformación genética vía Agrobacterium tumefaciens
Aunque existían resultados previos sobre la transformación genética de D. lanata con cepas silvestres de A. tumefaciens, el primer informe de la regeneración de plantas y el estudio de los transgenes en esta especie fue realizado por Lehmann et al. (1995). En este trabajo se emplearon dos vectores binarios, que contenían los genes nptII y uidA. La transformación genética se realizó con dos tipos de explante diferentes: discos foliares y protoplastos. En ambos casos se empleó como agente selectivo kanamicina a 80 μmol l-1 (~40 mg l-1). No se produjo formación de callos o de colonias celulares embriogénicas en los explantes no transformados, cultivados con el agente selectivo. Sin embargo, no fue posible obtener órganos o plantas a partir de los callos inducidos y multiplicados en selección. El cultivo de las colonias embriogénicas transformadas en medio de cultivo semisólido con el agente selectivo, permitió regenerar plantas vía embriogénesis somática. En contraste, las colonias transformadas seleccionadas en medio de cultivo líquido, tuvieron una baja formación de embriones resistentes. Las plantas regeneradas fueron similares a las no transformadas y no mostraron variaciones morfológicas asociadas a la presencia de los transgenes. Su integración en callos y plantas transformadas, fue comprobada por hibridación de Southern. Las actividades NPT-II y GUS, fueron variables de acuerdo con la naturaleza del explante analizado y el número de copias o la posición del transgén (Lehmann et al., 1995).
Este estudio demuestra que es posible transformar células de D. lanata con genes foráneos vía A. tumefaciens. Sin embargo, no se informaron las eficiencias de transformación y selección de los explantes infestados.
En 2002 se publicó un nuevo artículo sobre la transformación genética vía A. tumefaciens, esta vez en Digitalis minor L. Aunque el principal objetivo de estos investigadores fue proponer un protocolo de regeneración de esta especie, a partir de ápices caulinares y segmentos foliares (Sales et al., 2002). También realizaron aportes a esta temática y analizaron el contenido de cardenólidos de las plantas cultivadas in vitro, en comparación con muestras de plantas crecidas en casa de cultivo. Como parte de la investigación, explantes foliares y plántulas, fueron infestados con una cepa silvestre de A. tumefaciens y con un vector binario que contenía el gen uidA. En el primer caso se formaron tumores que se multiplicaron y posteriormente formaron raíces adventicias. Solo aquellos tumores que adquirieron una forma compacta y coloración verde, dieron origen a brotes (10-14%). Las plantas fueron más susceptibles a la infestación bacteriana en cuanto a la formación de tumores (93%), pero la formación de brotes tuvo una respuesta similar. La presencia de opinas fue detectada en todos los tumores analizados, no así en los brotes y raíces regenerados de estos.
Las explantes transformados con el vector binario, dieron lugar a resultados similares. La actividad GUS fue positiva en los tumores, pero no en los brotes analizados. Esto indicó que no se produjo una expresión estable de este gen. En un análisis mediante reacción en cadena de la polimerasa (PCR) de la presencia del gen uidA, solo fue posible observar el producto de la amplificación en cuatro de las ocho muestras analizadas. Las muestras positivas correspondían a los callos transformados y las negativas a los brotes regenerados de estos (Sales et al., 2002). El elevado número de explantes transformados en este estudio, en comparación con la baja eficiencia de transformación obtenida, es un ejemplo de la importancia del empleo de agentes selectivos para promover el crecimiento del tejido transformado y permitir al mismo tiempo el ahorro de recursos y mano de obra especializada.
Un año después ese grupo de trabajo publicó el primer informe de obtención de plantas transgénicas del género Digitalis, mediante la transformación genética de discos foliares con A. tumefaciens. Este trabajo también aportó nuevos conocimientos sobre el proceso de selección, pues además de los genes empleados en investigaciones anteriores, introdujeron el gen bar como marcador se selección (Sales et al., 2003).
En el citado estudio, luego del proceso de co-cultivo con la bacteria, los explantes fueron transferidos a medio de cultivo basal para la regeneración de plantas. En el caso de los transformados con el plásmido p35SGUSINT se adicionó al medio de cultivo kanamicina 35 mg l-1 o geneticina 10 mg l-1. Mientras, en los discos foliares transformados con pTAB16 se emplearon 2 mg l-1 de fosfinotricina para la selección. Estas concentraciones de agentes selectivos se determinaron en experimentos preliminares. Los explantes se mantuvieron en selección durante cuatro semanas, luego de las cuales los brotes fueron removidos y transferidos al mismo medio de cultivo de selección otros 14 días y luego transferidos para la elongación de 30-50 días. La integración de los transgenes en las plantas regeneradas fue confirmada por PCR e hibridación por Southern. También se realizaron ensayos GUS a los explantes.
Para la cepa bacteriana EHA105 la eficiencia de transformación sin acetosiringona, fue de solo un 0.5%. Esta pudo aumentarse al 16% al adicionar 100 μM de este compuesto fenólico. El 30% de esos explantes obtenidos luego de la transformación genética, contenían el gen nptII (38 plantas). La eficiencia de selección fue de 31% en todos los experimentos de transformación genética en los que se empleó la kanamicina como agente selectivo, lo cual implica que se produjo una alta ocurrencia de escapes (69%). Este resultado no es sorprendente, si se tiene en cuenta que en los experimentos anteriores en que se empleó este antibiótico, las concentraciones fueron superiores y también hubo escapes (Saito et al., 1990; Lehmann et al., 1995). Estas eficiencias de transformación genética y selección fueron modificadas cuando se varió la concentración de la bacteria, pero aún con presencia de escapes.
La eficiencia de transformación genética con la cepa AGL1, luego de la selección con fosfinotricina, fue muy baja (0.4-0.7%) y solo se obtuvieron tres plantas positivas. Sobre este resultado los autores sugirieron la hipótesis de que pudo deberse a la alta toxicidad de la fosfinotricina para D. minor, pero otros estudios son necesarios para confirmar este planteamiento. No se especifica si se producen escapes con este agente selectivo o con el empleo de geneticina. Sin embargo, proponen que la elevada incidencia de escapes se debe a la alta eficiencia de transformación en oposición a la baja de regeneración. Por lo cual se ejercería un efecto protector sobre las células no transformadas, al estar rodeadas de las que si contienen el GMS. Por último señalan que, aunque otros autores indican que la solución puede ser aumentar la concentración de kanamicina o emplear otro agente selectivo como la geneticina, ninguna de estas estrategias fueron efectivas en sus experimentos (Sales et al., 2003).
Las conclusiones de estos autores sobre el proceso de selección de plantas de D. minor en medio de cultivo de regeneración vía organogénesis directa, se corresponden con los aspectos anteriormente discutidos. La complejidad estructural del brote y el corto tiempo en que se produce la regeneración, impide que se realice una selección eficiente. Esto podría ser minimizado si se optimizan las condiciones de transformación genética. Pero, es de esperar que ocurran escapes siempre que se emplee un protocolo de este tipo.
En el 2007 Sales et al. publicaron el único trabajo conocido hasta el momento, en el cual se obtienen plantas transgénicas de D. minor que contienen un gen de interés. En este se introduce un gen codificante para el dominio catalítico de la 3-hidroxi-3-metilglutaril-CoA reductasa (EC 1.1.1.34), enzima clave de la ruta de síntesis del ácido mevalónico. Para ello, se empleó el protocolo propuesto por Sales et al. (2003) y el GMS nptII. Pero no se especifica el agente selectivo y la concentración empleada, ni las eficiencias de selección y transformación genética alcanzadas. Aunque no aporta nuevos elementos sobre el proceso de selección en la especie, demuestra que es posible modificar su metabolismo secundario y potenciar la producción de cardenólidos, mediante ingeniería genética (Sales et al., 2007). Para esto es necesario identificar las reacciones limitantes del proceso de síntesis de dichos compuestos y potenciar la actividad de las enzimas involucradas en su catálisis.
En 2008 y 2013 otro grupo de investigadores comenzaron a realizar nuevos estudios en Digitalis purpurea L. (Chong-Pérez et al., 2008; Kairuz et al., 2013). El objetivo del primer trabajo fue determinar la concentración mínima inhibitoria de geneticina (G418) en el proceso de formación de callos, para su posible uso como agente selectivo en la transformación genética de esta especie. En este informe se recomienda adicionar al medio de cultivo 50 mg l-1 de este agente selectivo con subcultivos cada 15 días (Chong-Pérez et al., 2008). La concentración seleccionada fue superior a las anteriormente empleadas en los trabajos de transformación genética de este género descritos, en los cuales se informaron escapes.
En el segundo trabajo también se selecciona la concentración mínima letal, pero esta vez con un antibiótico que no había sido empleado con anterioridad en Digitalis: higromicina B. Se realizaron en dos de los medios de cultivo utilizados en la regeneración indirecta de esta especie: formación de callos y multiplicación de brotes. Esto implica que se pueda mantener la presión selectiva durante todo el proceso de transformación genética y se minimice la aparición de escapes. Finalmente, se recomiendan las concentraciones de higromicina B 12 mg l-1 en medio de cultivo de formación de callos y de 75 mg l-1 en medio de cultivo de multiplicación de brotes, esta última luego de no menos de 30 días (Kairuz et al., 2013). En este trabajo se demostró que la higromicina B es más tóxica que la geneticina en iguales condiciones de selección y el GMS hpt podría ser más eficaz en la transformación genética de D. purpurea.
Aunque ambos estudios aportan nuevos conocimientos sobre el efecto de estos agentes selectivos, no son suficientes para conocer si la combinación GMS/agente selectivo es eficiente. Sería necesario realizar la transformación genética y determinar si existe diferenciación del tejido, crecimiento selectivo y si es posible regenerar plantas transgénicas genéticamente homogéneas, para poder afirmar que el esquema de selección empleado es eficiente.
Para el año 2014 se publicaron dos protocolos de transformación genética de segmentos foliares de D. purpurea, vía organogénesis directa (Li et al., 2014) e indirecta (Pérez-Alonso et al., 2014). En ambos casos se emplean los genes nptII y uidA, pero con diferentes cepas bacterianas y plásmidos. También difieren en cuanto al agente selectivo y, dado el protocolo de regeneración propuesto en cada caso, en las condiciones y tiempos de cultivo. Para ello, los autores establecen la composición del medio de cultivo y las condiciones de transformación genética (cepa bacteriana).
Li et al. (2014) adicionaron 100 mg l-1 de kanamicina al medio de cultivo para la inducción de brotes (4-6 semanas). Los explantes resistentes a kanamicina fueron transferidos a un medio de cultivo de enraizamiento (3-4 semanas), en el cual se mantiene la presión selectiva, pero con la mitad de la concentración del antibiótico. Dado que no se especifica, al parecer no se realizaron subcultivos para renovar el agente selectivo. Con las dos cepas bacterianas empleadas fue posible obtener una eficiencia de transformación genética de 52.2 y 60%, dado por el número de brotes resistentes a kanamicina que mostraron actividad GUS. Luego, los autores proponen un protocolo de transformación genética rápido y eficiente que podría ser empleado para estudiar las rutas de síntesis de cardenólidos.
En este trabajo se duplica la concentración de kanamicina que se emplea para la selección de segmentos foliares durante la inducción de brotes, en comparación con las publicaciones anteriormente analizadas. También se realiza la selección durante su enraizamiento, aunque en menor concentración, a pesar de que es un explante más complejo. Sin embargo, de igual modo a lo descrito por Sales et al. (2003) esto no imposibilita la aparición de escapes. Durante la discusión de los resultados los autores no abordaron este aspecto o las posibles causas que pudieron originarlo. Incluso no se tiene en cuenta la alta probabilidad de obtener plantas quiméricas, debido al origen pluricelular del brote y el agotamiento del antibiótico en el medio de cultivo. Por ello, aunque indudablemente es un protocolo que puede ser realizado en corto tiempo y con bajo costo, su eficiencia (0.6 líneas transgénicas por explante inicial) es discutible, si se tiene en cuenta que no se requiere de la realización de subcultivos y no se demuestra la homogeneidad genética de las líneas transgénicas. Tal vez, el empleo de un agente selectivo más tóxico para el tejido vegetal, cuya presión selectiva sea comparable a la capacidad de regeneración directa, podría minimizar estos riesgos. Nuevos estudios son necesarios para comprobar esta hipótesis.
Por otra parte, en el protocolo de transformación genética de segmentos foliares de D. purpurea vía organogénesis indirecta propuesto por Pérez-Alonso et al. (2014), se determinaron las condiciones de regeneración, la concentración mínima inhibitoria de geneticina (70 mg l-1) y se comparó la eficiencia de transformación genética para las cepas A. tumefaciens C58C1RifR (pMP90) y A. tumefaciens EHA105. En este caso el proceso de selección se realizó en medio de cultivo para la formación y multiplicación de callos, durante ocho semanas. Los callos resistentes a geneticina fueron seccionados y transferidos a medio de cultivo de regeneración sin antibiótico por igual tiempo. En ambos casos se realizaron subcultivos cada dos semanas.
La menor eficiencia de transformación genética se obtuvo con la cepa A. tumefaciens EHA105 (0.29). Fue posible alcanzar una media de 6.91 líneas transgénicas por explante inicial, cuando se empleó la cepa A. tumefaciens C58C1RifR (pMP90). El análisis histoquímico GUS mostró que el 58.5 y 62.3% de las plantas analizadas, fueron positivas. Estos autores proponen que la aparición de líneas GUS negativas pudo deberse a escapes del proceso selectivo, inserciones truncadas del ADN-T en el genoma de la planta o al silenciamiento del transgén. Las dos primeras justificaciones fueron comprobadas al realizar los análisis moleculares por PCR. Se detectó 4.6% de escapes del proceso selectivo, a pesar de lo cual es de cualquier modo la mayor eficiencia de selección informada en el género. Finalmente, puntualizaron que es el protocolo de transformación genética de D. purpurea vía A. tumefaciens más eficiente que se ha publicado hasta el momento y puede ser aplicado para la realización de estudios funcionales o en la obtención de nuevos genotipos con mayor producción de cardenólidos (Pérez-Alonso et al., 2014).
El resumen de todos los aspectos positivos y negativos de estos trabajos, posibilitará desarrollar nuevas estrategias de selección eficientes para la obtención de plantas transgénicas de Digitalis L.
CONCLUSIONES Y LÍNEAS DE INVESTIGACIÓN FUTURAS
El análisis de la literatura científica disponible, permite tomar decisiones en cuanto al diseño y la evaluación de esquemas de transformación genética y selección de eventos transgénicos en el género Digitalis. Primero debe realizarse la selección del GMS, determinar la concentración eficaz del agente selectivo, las condiciones de cultivo y por último la evaluación de su eficiencia. De acuerdo con la complejidad del explante, es recomendable mantener la presión selectiva en todos los medios de cultivo utilizados. Una estrategia de selección bien diseñada, puede llevar a la optimización de protocolos de transformación genética, el ahorro de recursos y a la obtención de plantas transgénicas genéticamente estables con caracteres agronómicos favorables.
Mucho queda por conocer en lo que se refiere a la transformación genética y la selección in vitro de plantas del género Digitalis. Una opción inmediata sería determinar la eficiencia de transformación genética con el gen hpt, del cual ya existen conocimientos básicos de su toxicidad en D. purpurea. También podría incluirse la implementación de nuevos esquemas de selección, ya sea con otros genes de selección positiva condicional, con GMS que no requieran el empleo de sustratos específicos, o con genes de selección negativa como codA.
Asimismo, el propio uso de GMS en la transformación genética de este cultivo, origina nuevos problemas científicos, como la necesidad de desarrollar una estrategia de escisión de marcadores de selección, según las diferentes metodologías descritas (Chong-Pérez y Angenon, 2013). Lo cual permitiría mejorar la percepción pública y la introducción en el mercado de los metabolitos resultantes de estas líneas genéticamente modificadas, para la potenciación de la síntesis de cardenólidos. Además podrían ser los primeros pasos para el desarrollo de estudios funcionales, empleando el sistema de edición genómica CRISPR/Cas (del inglés: Clustered Regularly Interspaced Short Palindromic Repeats/ CRISPR associated system).
Conflicto de intereses
Los autores no declaran conflicto de intereses.
REFERENCIAS
Bevan MW, Flavell RB, Chilton M-D (1983) A chimaeric antibiotic resistance gene as a selectable marker for plant cell transformation. Nature 304(5922): 184-187
Blochlinger K, Diggelmann H (1984) Hygromycin B phosphotransferase as a selectable marker for DNA transfer experiments with higher eucaryotic cells. Mol Cell Biol 4(12): 2929-31
Breyer D, Kopertekh L, Reheul D (2014) Alternatives to Antibiotic Resistance Marker Genes for In Vitro Selection of Genetically Modified Plants – Scientific Developments, Current Use, Operational Access and Biosafety Considerations. Critical Reviews in Plant Sciences 33(4): 286-330; doi:10.1080/07352689.2013.870422
Chong-Pérez B, Angenon G (2013) Strategies for Generating Marker-Free Transgenic Plants. En: Sithole-Niang I (Ed). Genetic Engineering, pp. 1-32. In Tech Publishing, Croacia
Chong-Pérez B, Kosky RG, Reyes M, Rojas L, Ocaña B, Tejeda M, Pérez B, Angenon G (2012) Heat shock induced excision of selectable marker genes in transgenic banana by the Cre-lox site-specific recombination system. Journal of Biotechnology 159(4): 265-273; doi:10.1016/j.jbiotec.2011.07.031
Chong-Pérez B, Pérez-Alonso N, Occeguera Z, Pérez-Capote A, Anabel P-P, Jiménez E (2008) Determinación de la concentración mínima inhibitoria de Geneticina G418 en el proceso de formación de callos de Digitalis purpurea L.. Biotecnología vegetal 8(2): 115-118
David B, Wolfender J-L, Dias DA (2015) The pharmaceutical industry and natural products: historical status and new trends. Phytochemistry Reviews 14(2): 299-315
de Oliveira MLP, Stover E, Thomson JG (2015) The codA gene as a negative selection marker in Citrus. SpringerPlus 4(1): 264-270; doi:10.1186/s40064-015-1047-y
Deliolanis NC, Ale A, Morscher S, Burton NC, Schaefer K, Radrich K, Razansky D, Ntziachristos V (2014) Deep-tissue reporter-gene imaging with fluorescence and optoacoustic tomography: a performance overview. Molecular imaging and biology 16(5): 652-660
Fraley RT, Rogers SG, Horsch RB, Sanders PR, Flick JS, Adams SP, Bittner ML (1983) Expression of bacterial genes in plant cells. Proceedings of the National Academy of Sciences 80(15): 4803-4807
Gallie DR, Lucas WJ, Walbot V (1989) Visualizing mRNA expression in plant protoplasts: factors influencing efficient mRNA uptake and translation. The Plant cell 1(3): 301-311
Gamborg OL, Miller RA, Ojima K (1968) Nutrient requirements of suspension cultures of soybean root cells. Experimental Cell Research 50(1): 151-158
Hanana M, Ayadi R, Mzid R, Khouja ML, Hanachi AS, Hamrouni L (2018) Efficient method of seed transformation via Agrobacterium tumefaciens for obtaining transgenic plants of Hibiscus cannabinus L.. Industrial Crops and Products 113(1): 274–282; doi:10.1016/j.indcrop.2018.01.050
Helmer G, Casadaban M, Bevan M, Kayes L, Chilton MD (1984) A new chimeric gene as a marker for plant transformation: The expression of Escherichia coli β-galactosidase in sunflower and tobacco cells. Bio/Technology 2(6): 520-527
Heron M, Anderson R (2016) Changes in the leading cause of death: Recent patterns in heart disease and cancer mortality. National Center for Health Statistics data brief (254): 1-8
Herrera-Estrella L, Block MD, Messens E, Hernalsteens JP, Montagu M V, Schell J (1983) Chimeric genes as dominant selectable markers in plant cells. The EMBO Journal. 2(6): 987-995
Jefferson RA, Kavanagh TA, Bevan MW (1987) GUS fusions: beta-glucuronidase as a sensitive and versatile gene fusion marker in higher plants. The EMBO journal 6(13): 3901-3907
Joersbo M, Okkels FT (1996) A novel principle for selection of transgenic plant cells: Positive selection. Plant Cell Reports 16(3-4): 219-221
Kairuz E, Pérez-Alonso N, Capote-Pérez A, Pérez-Pérez A, Jiménez E, Chong-Pérez B (2013) Concentración mínima letal de higromicina B en la formación de callos y multiplicación de brotes de Digitalis purpurea L.. Biotecnología Vegetal 13(1): 23-31
Kavita P, Burma PK (2008) A comparative analysis of green fluorescent protein and ß-glucuronidase protein-encoding genes as a reporter system for studying the temporal expression profiles of promoters. Journal of Biosciences 33(3): 337-343; doi:10.1007/s12038-008-0053-4
Kreis W, Muller-Uri F (2013) Cardenolide Aglycone Formation in Digitalis. En: Bach TJ, Rohmer M (Eds). Isoprenoid synthesis in plants and microorganisms: new concepts and experimental approaches, pp. 425-438. Springer Science + Business Media, New York
Lamont KC, Mudge SR, Liu G, Godwin ID (2017) Expression patterns of the native Shrunken-2 promoter in Sorghum bicolor visualised through use of the GFP reporter gene. Plant Cell Reports 36(11): 1689-1700; doi:10.1007/s00299-017-2182-4
Lehmann U, Moldenhauer D, Thomar S, Diettrich B, Luckner M (1995) Regeneration of Plants from Digitalis Lanata Cells Transformed with Agrobacterium tumefaciens Carrying Bacterial Genes Encoding Neomycin Phosphotransferase II and beta-glucuronidase. Journal of Plant Physiology 147(1): 53-57
Li Y, Gao Z, Piao C, Lu K, Wang Z, Cui ML (2014) A stable and efficient Agrobacterium tumefaciens-mediated genetic transformation of the medicinal plant Digitalis purpurea L.. Applied Biochemistry and Biotechnology 172(4): 1807-17; doi:10.1007/s12010-013-0648-6
Majhi BB, Bhosale R, Jawkar S, Veluthambi K (2014) Evaluation of codA, tms2, and ABRIN-A as negative selectable markers in transgenic tobacco and rice. In Vitro Cellular and Developmental Biology - Plant 50(5): 541-551; doi:10.1007/s11627-014-9625-1
Martín R, Chong-Pérez B, Pérez-Alonso N (2015) Organogénesis in vitro en el género Digitalis. Biotecnología Vegetal 15(4):195-206
Mathers CD, Stevens GA, Boerma T, White RA, Tobias MI (2015) Causes of international increases in older age life expectancy. The Lancet 385 (9967): 540-548
Miki B, Abdeen A, Manabe Y, Macdonald P (2009) Selectable marker genes and unintended changes to the plant transcriptome. Plant Biotechnology Journal 7: 211-218; doi:10.1111/j.1467-7652.2009.00400.x
Miki B, McHugh S (2004) Selectable marker genes in transgenic plants: applications, alternatives and biosafety. Journal of biotechnology 107(3): 193-232
Nietsch J, Brügmann T, Becker D, Fladung M (2017) Old methods rediscovered: application and improvement of two direct transformation methods to hybrid poplar (Populus tremula × P. alba). Plant Cell, Tissue and Organ Culture 130(1): 183-196
Osakabe K, Nishizawa-Yokoi A, Ohtsuki N, Osakabe Y, Toki S (2014) A mutated cytosine deaminase gene, codA (D314A), as an efficient negative selection marker for gene targeting in rice. Plant and Cell Physiology 55(3): 658-665; doi:10.1093/pcp/pct183
Padilla IMG, Burgos L (2010) Aminoglycoside antibiotics: Structure, functions and effects on in vitro plant culture and genetic transformation protocols. Plant Cell Reports 29: 1203-1213; doi:10.1007/s00299-010-0900-2
Parveez GKA, Majid NA (2018) Green fluorescent protein as a visual selection marker for oil palm transformation. Industrial Crops and Products 115(2): 134–145; doi:10.1016/j.indcrop.2018.02.016
Pérez-Alonso N, Chong-Pérez B, Capote A, Pérez A, Izquierdo Y, Angenon G, Jiménez E (2014) Agrobacterium tumefaciens-mediated genetic transformation of Digitalis purpurea L.. Plant Biotechnology Reports 8(5): 387-97; doi:10.1007/s11816-014-0329-0
Pérez-Alonso N, Wilken D, Gerth A, Jahn A, Nitzsche, Horst-Michael Kerns G, Capote-Pérez A, Jiménez E (2009) Cardiotonic glycosides from biomass of Digitalis purpurea L. cultured in temporary immersion systems. Plant Cell Tissue and Organ Culture 99(2): 151-156; doi:10.1007/s11240-009-9587-x
Pérez-Alonso NL, Arana LF, Capote PA, Pérez PA, Sosa R, Mollineda A, Jiménez E (2014) Estimulación de cardenólidos en brotes de Digitalis purpurea L. cultivados in vitro mediante elicitores. Revista Colombia de Biotecnologia 16(1): 51-61
Pérez-Alonso NL, Capote A, Gerth A, Jiménez E (2012) Increased cardenolides production by elicitation of Digitalis lanata shoots cultured in temporary immersion systems. Plant Cell Tissue and Organ Culture 110: 153-162; doi:10.1007/s11240-012-0139-4
Pradel H, Dumke-Lehmann U, Diettrich B, Luckner M (1997) Hairy root cultures of Digitalis lanata. Secondary metabolism and plant regeneration. Journal of Plant Physiology 151(2): 209-215
Prasher DC, Eckenrode VK, Ward WW, Prendergast FG, Cormier MJ (1992) Primary structure of the Aequorea victoria green-fluorescent protein. Gene 111(2): 229-33
Ramessar K, Peremarti A, Gómez-Galera S, Naqvi S, Moralejo M, Muñoz P, Capell T, Christou P (2007) Biosafety and risk assessment framework for selectable marker genes in transgenic crop plants: a case of the science not supporting the politics. Transgenic Research 16(3): 261-80; doi:10.1007/s11248-007-9083-1
Roca-Pérez L, Boluda R, Gavidia I, Pérez-Bermúdez P (2004) Seasonal cardenolide production and Dop5βr gene expression in natural populations of Digitalis obscura. Phytochemistry 65(13): 1869-78; doi:10.1016/j.phytochem.2004.05.004
Rosellini D (2011) Selectable marker genes from plants: Reliability and potential. In Vitro Cellular and Developmental Biology - Plant 47(2): 222-33; doi:10.1007/s11627-011-9348-5
Rosellini D (2012) Selectable Markers and Reporter Genes: A Well Furnished Toolbox for Plant Science and Genetic Engineering. Critical Reviews in Plant Sciences 31(5): 401-453; doi:10.1080/07352689.2012.683373
Saito K, Yamazaki M, Kaneko H, Murakoshi I, Fukuda Y, Van Montagu M (1991) Tissue-specific and stress-enhancing expression of the TR promoter for mannopine synthase in transgenic medicinal plants. Planta 184(1): 40-46
Saito K, Yamazaki M, Shimomura K, Yoshimatsu K, Murakoshi I (1990) Genetic transformation of foxglove (Digitalis purpurea) by chimeric foreign genes and production of cardioactive glycosides. Plant Cell Reports 9: 121-124
Sales E, Frieder M, Nebauer SG, Segura J, Kreis W, Arrillaga I (2011) Digitalis. En: Kole C (Ed). Wild Crop Relatives: Genomic and Breeding Resources, Plantation and Ornamental Crops, pp. 73-112. Heidelberg Springer-Verlag, Berlin
Sales E, Muñoz-Bertomeu J, Arrillaga I, Segura J (2007) Enhancement of Cardenolide and Phytosterol Levels by Expression of an N-Terminally Truncated 3-Hydroxy-3-methylglutaryl CoA Reductase in Transgenic Digitalis minor. Planta Medica 73(6): 605-610; doi:10.1055/s-2007-967199
Sales E, Nebauer SG, Arrillaga I, Segura J (2002) Plant hormones and Agrobacterium tumefaciens strain 82.139 induce efficient plant regeneration in the cardenolide-producing plant Digitalis minor. Journal of Plant Physiology 159(1): 9-16; doi:10.1078/0176-1617-00534
Sales E, Segura J, Arrillaga I (2003) Agrobacterium tumefaciens-Mediated Genetic Transformation of the Cardenolide-Producing Plant Digitalis minor L.. Planta Medica 69: 143-147
Shaner NC, Steinbach PA, Tsien RY (2005) A guide to choosing fluorescent proteins. Nature Methods 2(12): 905-9
Sheen J, Hwang S, Niwa Y, Kobayashi H, Galbraith DW (1995) Green‐fluorescent protein as a new vital marker in plant cells. The Plant Journal 8(5): 777-784
Stuhlemmer U, Kreis W (1996) Cardenolide formation and activity of pregnane-modifying enzymes in cell suspension cultures, shoot cultures and leaves of Digitalis lanata. Plant Physiology and Biochemistry 34(1): 85-91
Suzman R, Beard JR, Boerma T, Chatterji S (2015) Health in an ageing world - What do we know?. The Lancet 385(9967): 484-486; doi:10.1016/S0140-6736(14)61597-X
Verma SK, Das AK, Cingoz GS, Gurel E (2016) In vitro culture of Digitalis L. (Foxglove) and the production of cardenolides: An up-to-date review. Industrial Crops and Products 94: 20-51; doi:10.1016/j.indcrop.2016.08.031
Wakasa Y, Ozawa K, Takaiwa F (2007) Agrobacterium-mediated transformation of a low glutelin mutant of “Koshihikari” rice variety using the mutated-acetolactate synthase gene derived from rice genome as a selectable marker. Plant Cell Reports 26(9): 1567–1573; doi:10.1007/s00299-007-0373-0
Ziff OJ, Lane DA, Samra M, Griffith M, Kirchhof P, Lip GYH, Steeds RP, Townend J, Kotecha D (2015) Safety and efficacy of digoxin: systematic review and meta-analysis of observational and controlled trial data. BMJ (Clinical research ed) 351:h4451; doi:10.1136/bmj.h4451
Recibido: 24-01-2018
Aceptado: 01-03-2018
Copyright (c) 2018 Biotecnología Vegetal
Biotecnología Vegetal eISSN 2074-8647, RNPS: 2154. ISSN 1609-1841, RNPS: 0397 Editada por: Instituto de Biotecnología de las Plantas. Universidad Central Marta Abreu de Las Villas. Carretera a Camajuaní km 5.5, Santa Clara, Villa Clara, Cuba CP 54 830 Tel: 53 42200124, e-mail: info@ibp.co.cu

Biotecnología Vegetal está bajo una Licencia Creative Commons Atribución-NoComercial 4.0 Internacional.
